ホーム » sales-info の投稿
作者アーカイブ: sales-info
MONAI 1.0 : tutorials : 3D セグメンテーション – 脾臓 3D セグメンテーション
MONAI 1.0 : tutorials : 3D セグメンテーション – 脾臓 3D セグメンテーション (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 01/19/2023 (1.1.0)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP

MONAI 0.7 : tutorials : 3D セグメンテーション – 脾臓 3D セグメンテーション
このノートブックは MSD 脾臓データセット に基づいた 3D セグメンテーションの end-to-end な訓練と評価サンプルです。このサンプルは PyTorch ベースのプログラムで MONAI モジュールの柔軟性を示します :
- 辞書ベースの訓練データ構造のための変換。
- メタデータを含む NIfTI 画像をロードする。
- 想定する範囲で医用画像強度をスケールする。
- ポジティブ/ネガティブ・ラベル比率に基づいてバランスの取れた画像パッチサンプルのバッチをクロップする。
- 訓練と検証を高速化するキャッシュ IO と変換。
- 3D セグメンテーション・タスクのための 3D UNet, Dice 損失関数, Mean Dice メトリック。
- スライディング・ウィンドウ推論。
- 再現性のための決定論的訓練。
このチュートリアルは MONAI を既存の PyTorch 医用 DL プログラムに統合する方法を示します。
そして以下の機能を簡単に使用することができます :
- 辞書形式データのための変換。
- メタデータを含む Nifti 画像をロードする。
- チャネル次元がない場合チャネル dim をデータに追加する。
- 想定される範囲で医用画像強度をスケールする。
- ポジティブ/ネガティブ・ラベル比率に基づいてバランスの取れた画像のバッチをクロップする。
- 訓練と検証を高速化するキャシュ IO と変換。
- 3D セグメンテーション・タスクのための 3D UNet モデル、Dice 損失関数、Mean Dice メトリック。
- スライディング・ウィンドウ推論手法。
- 再現性のための決定論的訓練。
Spleen データセットは http://medicaldecathlon.com/ からダウンロードできます。

- Target: Spleen
- Modality: CT
- Size: 61 3D volumes (41 Training + 20 Testing)
- Source: Memorial Sloan Kettering Cancer Center
- Challenge: Large ranging foreground size
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[gdown, nibabel, tqdm, ignite]"
!python -c "import matplotlib" || pip install -q matplotlib
%matplotlib inline
インポートのセットアップ
from monai.utils import first, set_determinism
from monai.transforms import (
AsDiscrete,
AsDiscreted,
EnsureChannelFirstd,
Compose,
CropForegroundd,
LoadImaged,
Orientationd,
RandCropByPosNegLabeld,
SaveImaged,
ScaleIntensityRanged,
Spacingd,
Invertd,
)
from monai.handlers.utils import from_engine
from monai.networks.nets import UNet
from monai.networks.layers import Norm
from monai.metrics import DiceMetric
from monai.losses import DiceLoss
from monai.inferers import sliding_window_inference
from monai.data import CacheDataset, DataLoader, Dataset, decollate_batch
from monai.config import print_config
from monai.apps import download_and_extract
import torch
import matplotlib.pyplot as plt
import tempfile
import shutil
import os
import glob
print_config()
MONAI version: 1.1.0+2.g97918e46
Numpy version: 1.22.2
Pytorch version: 1.13.0a0+d0d6b1f
MONAI flags: HAS_EXT = True, USE_COMPILED = False, USE_META_DICT = False
MONAI rev id: 97918e46e0d2700c050e678d72e3edb35afbd737
MONAI __file__: /opt/monai/monai/__init__.py
Optional dependencies:
Pytorch Ignite version: 0.4.10
Nibabel version: 4.0.2
scikit-image version: 0.19.3
Pillow version: 9.0.1
Tensorboard version: 2.10.1
gdown version: 4.6.0
TorchVision version: 0.14.0a0
tqdm version: 4.64.1
lmdb version: 1.3.0
psutil version: 5.9.2
pandas version: 1.4.4
einops version: 0.6.0
transformers version: 4.21.3
mlflow version: 2.0.1
pynrrd version: 1.0.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。
これは結果をセーブしてダウンロードを再利用することを可能にします。
指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical/
データセットのダウンロード
データセットをダウンロードして展開します。データセットは http://medicaldecathlon.com/ に由来します。
resource = "https://msd-for-monai.s3-us-west-2.amazonaws.com/Task09_Spleen.tar"
md5 = "410d4a301da4e5b2f6f86ec3ddba524e"
compressed_file = os.path.join(root_dir, "Task09_Spleen.tar")
data_dir = os.path.join(root_dir, "Task09_Spleen")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
MSD 脾臓データセット・パスの設定
train_images = sorted(
glob.glob(os.path.join(data_dir, "imagesTr", "*.nii.gz")))
train_labels = sorted(
glob.glob(os.path.join(data_dir, "labelsTr", "*.nii.gz")))
data_dicts = [
{"image": image_name, "label": label_name}
for image_name, label_name in zip(train_images, train_labels)
]
train_files, val_files = data_dicts[:-9], data_dicts[-9:]
再現性のための決定論的訓練の設定
set_determinism(seed=0)
訓練と検証のための変換のセットアップ
ここではデータセットを増強するために幾つかの変換を使用します :
- LoadImaged は NIfTI 形式ファイルから脾臓 CT 画像とラベルをロードします。
- EnsureChannelFirstd は元のデータが「チャネル first」shape を構成することを保証します。
- Orientationd はアフィン行列に基づいてデータの向きを統一します。
- Spacingd はアフィン行列に基づいて pixdim=(1.5, 1.5, 2.) による spacing を調整します。
- ScaleIntensityRanged は強度範囲 [-57, 164] を抽出して [0, 1] にスケールします。
- CropForegroundd は画像とラベルの valid body 領域にフォーカスするために総てのゼロ境界 (= border) を削除します。
- RandCropByPosNegLabeld は pos / neg 比率に基づいて大きな画像からランダムにパッチサンプルをクロップします。
ネガティブサンプルの画像中心は valid body 領域になければなりません。
- RandAffined は PyTorch アフィン変換に基づいて回転, スケール, shear, 並行移動等を一緒に効率的に実行します。
train_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys=["image", "label"]),
ScaleIntensityRanged(
keys=["image"], a_min=-57, a_max=164,
b_min=0.0, b_max=1.0, clip=True,
),
CropForegroundd(keys=["image", "label"], source_key="image"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(keys=["image", "label"], pixdim=(
1.5, 1.5, 2.0), mode=("bilinear", "nearest")),
RandCropByPosNegLabeld(
keys=["image", "label"],
label_key="label",
spatial_size=(96, 96, 96),
pos=1,
neg=1,
num_samples=4,
image_key="image",
image_threshold=0,
),
# user can also add other random transforms
# RandAffined(
# keys=['image', 'label'],
# mode=('bilinear', 'nearest'),
# prob=1.0, spatial_size=(96, 96, 96),
# rotate_range=(0, 0, np.pi/15),
# scale_range=(0.1, 0.1, 0.1)),
]
)
val_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys=["image", "label"]),
ScaleIntensityRanged(
keys=["image"], a_min=-57, a_max=164,
b_min=0.0, b_max=1.0, clip=True,
),
CropForegroundd(keys=["image", "label"], source_key="image"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(keys=["image", "label"], pixdim=(
1.5, 1.5, 2.0), mode=("bilinear", "nearest")),
]
)
DataLoaer で変換を確認する
check_ds = Dataset(data=val_files, transform=val_transforms)
check_loader = DataLoader(check_ds, batch_size=1)
check_data = first(check_loader)
image, label = (check_data["image"][0][0], check_data["label"][0][0])
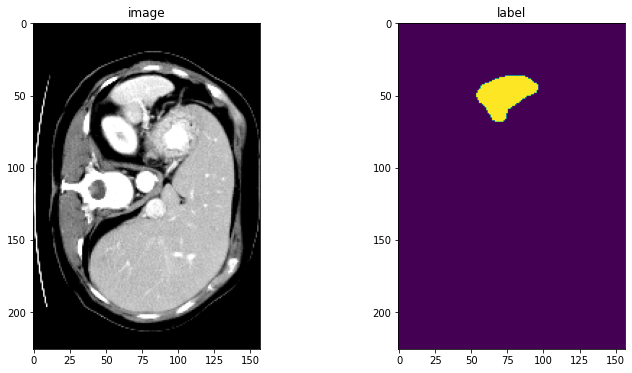
print(f"image shape: {image.shape}, label shape: {label.shape}")
# plot the slice [:, :, 80]
plt.figure("check", (12, 6))
plt.subplot(1, 2, 1)
plt.title("image")
plt.imshow(image[:, :, 80], cmap="gray")
plt.subplot(1, 2, 2)
plt.title("label")
plt.imshow(label[:, :, 80])
plt.show()
image shape: torch.Size([226, 157, 113]), label shape: torch.Size([226, 157, 113])
訓練と検証のために CacheDataset と DataLoader を定義する
ここで訓練と検証プロセスを高速化するために CacheDataset を使用し、それは通常の Dataset よりも 10x 高速です。ベストなパフォーマンスを得るためには、総てのデータをキャッシュするために cache_rate=1.0 を設定します、メモリが十分でない場合には、低い値を設定してください。ユーザはまた cache_rate の代わりに cache_num を設定することもできて、2 つの設定の最小値を使用します。そしてキャッシュする間にマルチスレッドを有効にするために num_workers を設定します。通常の Dataset を試したい場合は、下でコメントされたコードを単に使用するように変更してください。
train_ds = CacheDataset(
data=train_files, transform=train_transforms,
cache_rate=1.0, num_workers=4)
# train_ds = Dataset(data=train_files, transform=train_transforms)
# use batch_size=2 to load images and use RandCropByPosNegLabeld
# to generate 2 x 4 images for network training
train_loader = DataLoader(train_ds, batch_size=2, shuffle=True, num_workers=4)
val_ds = CacheDataset(
data=val_files, transform=val_transforms, cache_rate=1.0, num_workers=4)
# val_ds = Dataset(data=val_files, transform=val_transforms)
val_loader = DataLoader(val_ds, batch_size=1, num_workers=4)
Loading dataset: 100%|██████████| 32/32 [00:32<00:00, 1.02s/it] Loading dataset: 100%|██████████| 9/9 [00:07<00:00, 1.18it/s]
モデル、損失、Optimizer を作成する
# standard PyTorch program style: create UNet, DiceLoss and Adam optimizer
device = torch.device("cuda:0")
model = UNet(
spatial_dims=3,
in_channels=1,
out_channels=2,
channels=(16, 32, 64, 128, 256),
strides=(2, 2, 2, 2),
num_res_units=2,
norm=Norm.BATCH,
).to(device)
loss_function = DiceLoss(to_onehot_y=True, softmax=True)
optimizer = torch.optim.Adam(model.parameters(), 1e-4)
dice_metric = DiceMetric(include_background=False, reduction="mean")
典型的な PyTorch 訓練プロセスを実行する
max_epochs = 600
val_interval = 2
best_metric = -1
best_metric_epoch = -1
epoch_loss_values = []
metric_values = []
post_pred = Compose([AsDiscrete(argmax=True, to_onehot=2)])
post_label = Compose([AsDiscrete(to_onehot=2)])
for epoch in range(max_epochs):
print("-" * 10)
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step += 1
inputs, labels = (
batch_data["image"].to(device),
batch_data["label"].to(device),
)
optimizer.zero_grad()
outputs = model(inputs)
loss = loss_function(outputs, labels)
loss.backward()
optimizer.step()
epoch_loss += loss.item()
print(
f"{step}/{len(train_ds) // train_loader.batch_size}, "
f"train_loss: {loss.item():.4f}")
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
if (epoch + 1) % val_interval == 0:
model.eval()
with torch.no_grad():
for val_data in val_loader:
val_inputs, val_labels = (
val_data["image"].to(device),
val_data["label"].to(device),
)
roi_size = (160, 160, 160)
sw_batch_size = 4
val_outputs = sliding_window_inference(
val_inputs, roi_size, sw_batch_size, model)
val_outputs = [post_pred(i) for i in decollate_batch(val_outputs)]
val_labels = [post_label(i) for i in decollate_batch(val_labels)]
# compute metric for current iteration
dice_metric(y_pred=val_outputs, y=val_labels)
# aggregate the final mean dice result
metric = dice_metric.aggregate().item()
# reset the status for next validation round
dice_metric.reset()
metric_values.append(metric)
if metric > best_metric:
best_metric = metric
best_metric_epoch = epoch + 1
torch.save(model.state_dict(), os.path.join(
root_dir, "best_metric_model.pth"))
print("saved new best metric model")
print(
f"current epoch: {epoch + 1} current mean dice: {metric:.4f}"
f"\nbest mean dice: {best_metric:.4f} "
f"at epoch: {best_metric_epoch}"
)
print(
f"train completed, best_metric: {best_metric:.4f} "
f"at epoch: {best_metric_epoch}")
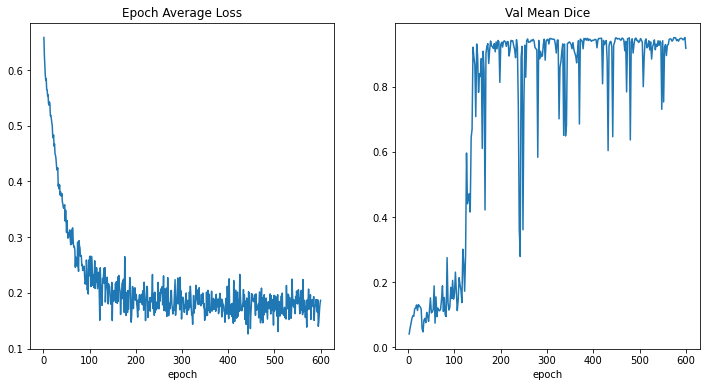
train completed, best_metric: 0.9510 at epoch: 598
損失とメトリックをプロットする
plt.figure("train", (12, 6))
plt.subplot(1, 2, 1)
plt.title("Epoch Average Loss")
x = [i + 1 for i in range(len(epoch_loss_values))]
y = epoch_loss_values
plt.xlabel("epoch")
plt.plot(x, y)
plt.subplot(1, 2, 2)
plt.title("Val Mean Dice")
x = [val_interval * (i + 1) for i in range(len(metric_values))]
y = metric_values
plt.xlabel("epoch")
plt.plot(x, y)
plt.show()
入力画像とラベルでベストなモデル出力を確認する
model.load_state_dict(torch.load(
os.path.join(root_dir, "best_metric_model.pth")))
model.eval()
with torch.no_grad():
for i, val_data in enumerate(val_loader):
roi_size = (160, 160, 160)
sw_batch_size = 4
val_outputs = sliding_window_inference(
val_data["image"].to(device), roi_size, sw_batch_size, model
)
# plot the slice [:, :, 80]
plt.figure("check", (18, 6))
plt.subplot(1, 3, 1)
plt.title(f"image {i}")
plt.imshow(val_data["image"][0, 0, :, :, 80], cmap="gray")
plt.subplot(1, 3, 2)
plt.title(f"label {i}")
plt.imshow(val_data["label"][0, 0, :, :, 80])
plt.subplot(1, 3, 3)
plt.title(f"output {i}")
plt.imshow(torch.argmax(
val_outputs, dim=1).detach().cpu()[0, :, :, 80])
plt.show()
if i == 2:
break



元の画像 spacing 上の評価
val_org_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys=["image", "label"]),
Orientationd(keys=["image"], axcodes="RAS"),
Spacingd(keys=["image"], pixdim=(
1.5, 1.5, 2.0), mode="bilinear"),
ScaleIntensityRanged(
keys=["image"], a_min=-57, a_max=164,
b_min=0.0, b_max=1.0, clip=True,
),
CropForegroundd(keys=["image"], source_key="image"),
]
)
val_org_ds = Dataset(
data=val_files, transform=val_org_transforms)
val_org_loader = DataLoader(val_org_ds, batch_size=1, num_workers=4)
post_transforms = Compose([
Invertd(
keys="pred",
transform=val_org_transforms,
orig_keys="image",
meta_keys="pred_meta_dict",
orig_meta_keys="image_meta_dict",
meta_key_postfix="meta_dict",
nearest_interp=False,
to_tensor=True,
device="cpu",
),
AsDiscreted(keys="pred", argmax=True, to_onehot=2),
AsDiscreted(keys="label", to_onehot=2),
])
model.load_state_dict(torch.load(
os.path.join(root_dir, "best_metric_model.pth")))
model.eval()
with torch.no_grad():
for val_data in val_org_loader:
val_inputs = val_data["image"].to(device)
roi_size = (160, 160, 160)
sw_batch_size = 4
val_data["pred"] = sliding_window_inference(
val_inputs, roi_size, sw_batch_size, model)
val_data = [post_transforms(i) for i in decollate_batch(val_data)]
val_outputs, val_labels = from_engine(["pred", "label"])(val_data)
# compute metric for current iteration
dice_metric(y_pred=val_outputs, y=val_labels)
# aggregate the final mean dice result
metric_org = dice_metric.aggregate().item()
# reset the status for next validation round
dice_metric.reset()
print("Metric on original image spacing: ", metric_org)
Metric on original image spacing: 0.9637420177459717
テストセット上の推論
test_images = sorted(
glob.glob(os.path.join(data_dir, "imagesTs", "*.nii.gz")))
test_data = [{"image": image} for image in test_images]
test_org_transforms = Compose(
[
LoadImaged(keys="image"),
EnsureChannelFirstd(keys="image"),
Orientationd(keys=["image"], axcodes="RAS"),
Spacingd(keys=["image"], pixdim=(
1.5, 1.5, 2.0), mode="bilinear"),
ScaleIntensityRanged(
keys=["image"], a_min=-57, a_max=164,
b_min=0.0, b_max=1.0, clip=True,
),
CropForegroundd(keys=["image"], source_key="image"),
]
)
test_org_ds = Dataset(
data=test_data, transform=test_org_transforms)
test_org_loader = DataLoader(test_org_ds, batch_size=1, num_workers=4)
post_transforms = Compose([
Invertd(
keys="pred",
transform=test_org_transforms,
orig_keys="image",
meta_keys="pred_meta_dict",
orig_meta_keys="image_meta_dict",
meta_key_postfix="meta_dict",
nearest_interp=False,
to_tensor=True,
),
AsDiscreted(keys="pred", argmax=True, to_onehot=2),
SaveImaged(keys="pred", meta_keys="pred_meta_dict", output_dir="./out", output_postfix="seg", resample=False),
])
# # uncomment the following lines to visualize the predicted results
# from monai.transforms import LoadImage
# loader = LoadImage()
model.load_state_dict(torch.load(
os.path.join(root_dir, "best_metric_model.pth")))
model.eval()
with torch.no_grad():
for test_data in test_org_loader:
test_inputs = test_data["image"].to(device)
roi_size = (160, 160, 160)
sw_batch_size = 4
test_data["pred"] = sliding_window_inference(
test_inputs, roi_size, sw_batch_size, model)
test_data = [post_transforms(i) for i in decollate_batch(test_data)]
# # uncomment the following lines to visualize the predicted results
# test_output = from_engine(["pred"])(test_data)
# original_image = loader(test_output[0].meta["filename_or_obj"])
# plt.figure("check", (18, 6))
# plt.subplot(1, 2, 1)
# plt.imshow(original_image[:, :, 20], cmap="gray")
# plt.subplot(1, 2, 2)
# plt.imshow(test_output[0].detach().cpu()[1, :, :, 20])
# plt.show()
データディレクトリのクリーンアップ
一時ディレクトリが使用された場合にはディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
MONAI 1.0 : tutorials : 3D セグメンテーション – 脳腫瘍 3D セグメンテーション
MONAI 1.0 : tutorials : 3D セグメンテーション – 脳腫瘍 3D セグメンテーション (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 01/18/2023 (1.1.0)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP
MONAI 1.0 : tutorials : 3D セグメンテーション – 脳腫瘍 3D セグメンテーション
このチュートリアルは MSD 脳腫瘍データセット に基づくマルチラベル・セグメンテーションタスクの訓練ワークフローを構築する方法を示します。
このチュートリアルはマルチラベル・セグメンテーション・タスクの訓練ワークフローを構築する方法を示します。
そしてそれは以下の機能を含みます :
- 辞書形式データ用の変換。
- MONAI 変換 API に従って新しい変換を定義する。
- メタデータと共に Nifti 画像をロードし、画像のリストをロードしてそれらをスタックする。
- データ増強のために強度をランダムに調整する。
- 訓練と検証を高速化する Cache IO と変換。
- 3D セグメンテーション・タスクのための 3D SegResNet モデル, Dice 損失関数, 平均 Dice メトリック。
- 再現性のための決定論的訓練。
データセットは http://medicaldecathlon.com/ に由来します。
ターゲット: Gliomas segmentation necrotic/active tumour and oedema
モダリティ: マルチモーダル multisite MRI データ (FLAIR, T1w, T1gd,T2w)
サイズ: 750 4D ボリューム (484 訓練 + 266 テスト)
ソース: BRATS 2016 と 2017 データセット。
チャレンジ: Complex and heterogeneously-located targets
下図は、様々なモダリティでアノテートされている腫瘍部分領域の画像パッチ (左上) とデータセット全体のための最終的なラベル (右) を示します。(図は BraTS IEEE TMI 論文 から引用)
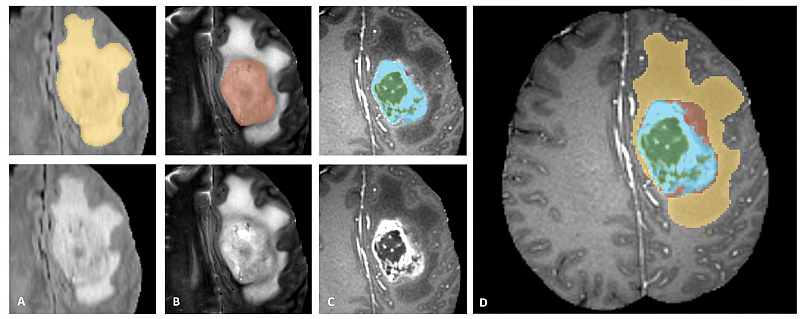
画像パッチは左から右へ以下を示します :
- T2-FLAIR で見える腫瘍全体 (黄色) (Fig.A)。
- T2 で見える腫瘍のコア (赤色) (Fig. B)。
- T1Gd で見える enhancing 腫瘍構造 (ライトブルー)、これはコアの嚢胞 (のうほう) (= cystic) / 壊死 (=necrotic) 成分 (緑色) を取り囲んでいます (Fig. C)。
- セグメンテーションは 腫瘍部分領域の最終的なラベル (Fig.D) を生成するために組み合わされます : 浮腫 (= edema) (黄色), non-enhancing ソリッドコア (赤色), 嚢胞/壊死コア (緑色), enhancing コア (青色) です。
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[nibabel, tqdm]"
!python -c "import matplotlib" || pip install -q matplotlib
%matplotlib inline
インポートのセットアップ
import os
import shutil
import tempfile
import time
import matplotlib.pyplot as plt
from monai.apps import DecathlonDataset
from monai.config import print_config
from monai.data import DataLoader, decollate_batch
from monai.handlers.utils import from_engine
from monai.losses import DiceLoss
from monai.inferers import sliding_window_inference
from monai.metrics import DiceMetric
from monai.networks.nets import SegResNet
from monai.transforms import (
Activations,
Activationsd,
AsDiscrete,
AsDiscreted,
Compose,
Invertd,
LoadImaged,
MapTransform,
NormalizeIntensityd,
Orientationd,
RandFlipd,
RandScaleIntensityd,
RandShiftIntensityd,
RandSpatialCropd,
Spacingd,
EnsureTyped,
EnsureChannelFirstd,
)
from monai.utils import set_determinism
import torch
print_config()
MONAI version: 0.9.1
Numpy version: 1.22.4
Pytorch version: 1.13.0a0+340c412
MONAI flags: HAS_EXT = True, USE_COMPILED = False, USE_META_DICT = False
MONAI rev id: 356d2d2f41b473f588899d705bbc682308cee52c
MONAI __file__: /opt/monai/monai/__init__.py
Optional dependencies:
Pytorch Ignite version: 0.4.9
Nibabel version: 4.0.1
scikit-image version: 0.19.3
Pillow version: 9.0.1
Tensorboard version: 2.9.1
gdown version: 4.5.1
TorchVision version: 0.13.0a0
tqdm version: 4.64.0
lmdb version: 1.3.0
psutil version: 5.9.1
pandas version: 1.3.5
einops version: 0.4.1
transformers version: 4.20.1
mlflow version: 1.27.0
pynrrd version: 0.4.3
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。これは結果をセーブしてダウンロードを再利用することを可能にします。指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical
再現性のために決定論的訓練を設定する
set_determinism(seed=0)
脳腫瘍のラベルを変換するために新しい変換を定義する
ここでは多クラスラベルを One-Hot 形式のマルチラベルのセグメンテーション・タスクに変換します。
class ConvertToMultiChannelBasedOnBratsClassesd(MapTransform):
"""
Convert labels to multi channels based on brats classes:
label 1 is the peritumoral edema
label 2 is the GD-enhancing tumor
label 3 is the necrotic and non-enhancing tumor core
The possible classes are TC (Tumor core), WT (Whole tumor)
and ET (Enhancing tumor).
"""
def __call__(self, data):
d = dict(data)
for key in self.keys:
result = []
# merge label 2 and label 3 to construct TC
result.append(torch.logical_or(d[key] == 2, d[key] == 3))
# merge labels 1, 2 and 3 to construct WT
result.append(
torch.logical_or(
torch.logical_or(d[key] == 2, d[key] == 3), d[key] == 1
)
)
# label 2 is ET
result.append(d[key] == 2)
d[key] = torch.stack(result, axis=0).float()
return d
訓練と検証のための変換のセットアップ
train_transform = Compose(
[
# load 4 Nifti images and stack them together
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
EnsureTyped(keys=["image", "label"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
RandSpatialCropd(keys=["image", "label"], roi_size=[224, 224, 144], random_size=False),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=0),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=1),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=2),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
RandScaleIntensityd(keys="image", factors=0.1, prob=1.0),
RandShiftIntensityd(keys="image", offsets=0.1, prob=1.0),
]
)
val_transform = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
EnsureTyped(keys=["image", "label"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image", "label"], axcodes="RAS"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
]
)
DecathlonDataset でデータを素早くロードする
ここではデータセットを自動的にダウンロードして抽出するために DecathlonDataset を使用します。それは MONAI CacheDataset を継承し、より少ないメモリを使用したい場合には、訓練のために N 項目をキャッシュするために cache_num=N を設定して検証のために総ての項目をキャッシュするために default args を使用できます、それはメモリサイズに依存します。
# here we don't cache any data in case out of memory issue
train_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=train_transform,
section="training",
download=True,
cache_rate=0.0,
num_workers=4,
)
train_loader = DataLoader(train_ds, batch_size=1, shuffle=True, num_workers=4)
val_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="validation",
download=False,
cache_rate=0.0,
num_workers=4,
)
val_loader = DataLoader(val_ds, batch_size=1, shuffle=False, num_workers=4)
Verified 'Task01_BrainTumour.tar', md5: 240a19d752f0d9e9101544901065d872. File exists: /workspace/data/medical/Task01_BrainTumour.tar, skipped downloading. Non-empty folder exists in /workspace/data/medical/Task01_BrainTumour, skipped extracting.
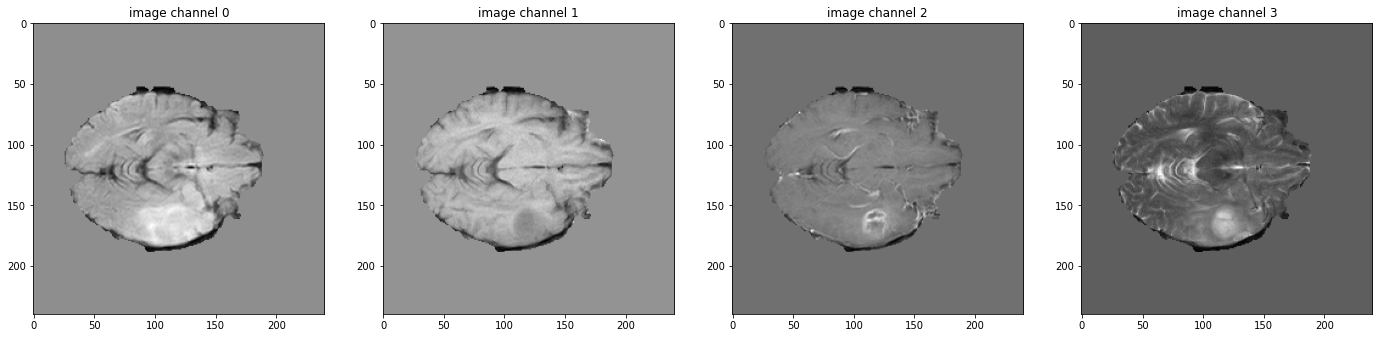
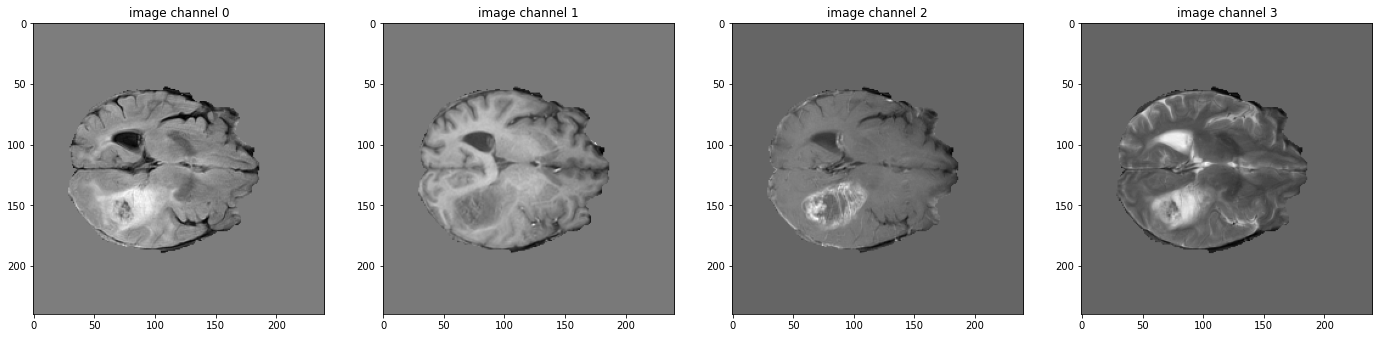
データ shape を確認して可視化する
# pick one image from DecathlonDataset to visualize and check the 4 channels
val_data_example = val_ds[2]
print(f"image shape: {val_data_example['image'].shape}")
plt.figure("image", (24, 6))
for i in range(4):
plt.subplot(1, 4, i + 1)
plt.title(f"image channel {i}")
plt.imshow(val_data_example["image"][i, :, :, 60].detach().cpu(), cmap="gray")
plt.show()
# also visualize the 3 channels label corresponding to this image
print(f"label shape: {val_data_example['label'].shape}")
plt.figure("label", (18, 6))
for i in range(3):
plt.subplot(1, 3, i + 1)
plt.title(f"label channel {i}")
plt.imshow(val_data_example["label"][i, :, :, 60].detach().cpu())
plt.show()
image shape: torch.Size([4, 240, 240, 155])

モデル, 損失, Optimizer を作成する
max_epochs = 300
val_interval = 1
VAL_AMP = True
# standard PyTorch program style: create SegResNet, DiceLoss and Adam optimizer
device = torch.device("cuda:0")
model = SegResNet(
blocks_down=[1, 2, 2, 4],
blocks_up=[1, 1, 1],
init_filters=16,
in_channels=4,
out_channels=3,
dropout_prob=0.2,
).to(device)
loss_function = DiceLoss(smooth_nr=0, smooth_dr=1e-5, squared_pred=True, to_onehot_y=False, sigmoid=True)
optimizer = torch.optim.Adam(model.parameters(), 1e-4, weight_decay=1e-5)
lr_scheduler = torch.optim.lr_scheduler.CosineAnnealingLR(optimizer, T_max=max_epochs)
dice_metric = DiceMetric(include_background=True, reduction="mean")
dice_metric_batch = DiceMetric(include_background=True, reduction="mean_batch")
post_trans = Compose(
[Activations(sigmoid=True), AsDiscrete(threshold=0.5)]
)
# define inference method
def inference(input):
def _compute(input):
return sliding_window_inference(
inputs=input,
roi_size=(240, 240, 160),
sw_batch_size=1,
predictor=model,
overlap=0.5,
)
if VAL_AMP:
with torch.cuda.amp.autocast():
return _compute(input)
else:
return _compute(input)
# use amp to accelerate training
scaler = torch.cuda.amp.GradScaler()
# enable cuDNN benchmark
torch.backends.cudnn.benchmark = True
典型的な PyTorch 訓練プロセスの実行
best_metric = -1
best_metric_epoch = -1
best_metrics_epochs_and_time = [[], [], []]
epoch_loss_values = []
metric_values = []
metric_values_tc = []
metric_values_wt = []
metric_values_et = []
total_start = time.time()
for epoch in range(max_epochs):
epoch_start = time.time()
print("-" * 10)
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step_start = time.time()
step += 1
inputs, labels = (
batch_data["image"].to(device),
batch_data["label"].to(device),
)
optimizer.zero_grad()
with torch.cuda.amp.autocast():
outputs = model(inputs)
loss = loss_function(outputs, labels)
scaler.scale(loss).backward()
scaler.step(optimizer)
scaler.update()
epoch_loss += loss.item()
print(
f"{step}/{len(train_ds) // train_loader.batch_size}"
f", train_loss: {loss.item():.4f}"
f", step time: {(time.time() - step_start):.4f}"
)
lr_scheduler.step()
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
if (epoch + 1) % val_interval == 0:
model.eval()
with torch.no_grad():
for val_data in val_loader:
val_inputs, val_labels = (
val_data["image"].to(device),
val_data["label"].to(device),
)
val_outputs = inference(val_inputs)
val_outputs = [post_trans(i) for i in decollate_batch(val_outputs)]
dice_metric(y_pred=val_outputs, y=val_labels)
dice_metric_batch(y_pred=val_outputs, y=val_labels)
metric = dice_metric.aggregate().item()
metric_values.append(metric)
metric_batch = dice_metric_batch.aggregate()
metric_tc = metric_batch[0].item()
metric_values_tc.append(metric_tc)
metric_wt = metric_batch[1].item()
metric_values_wt.append(metric_wt)
metric_et = metric_batch[2].item()
metric_values_et.append(metric_et)
dice_metric.reset()
dice_metric_batch.reset()
if metric > best_metric:
best_metric = metric
best_metric_epoch = epoch + 1
best_metrics_epochs_and_time[0].append(best_metric)
best_metrics_epochs_and_time[1].append(best_metric_epoch)
best_metrics_epochs_and_time[2].append(time.time() - total_start)
torch.save(
model.state_dict(),
os.path.join(root_dir, "best_metric_model.pth"),
)
print("saved new best metric model")
print(
f"current epoch: {epoch + 1} current mean dice: {metric:.4f}"
f" tc: {metric_tc:.4f} wt: {metric_wt:.4f} et: {metric_et:.4f}"
f"\nbest mean dice: {best_metric:.4f}"
f" at epoch: {best_metric_epoch}"
)
print(f"time consuming of epoch {epoch + 1} is: {(time.time() - epoch_start):.4f}")
total_time = time.time() - total_start
print(f"train completed, best_metric: {best_metric:.4f} at epoch: {best_metric_epoch}, total time: {total_time}.")
train completed, best_metric: 0.7914 at epoch: 279, total time: 90155.70936012268.
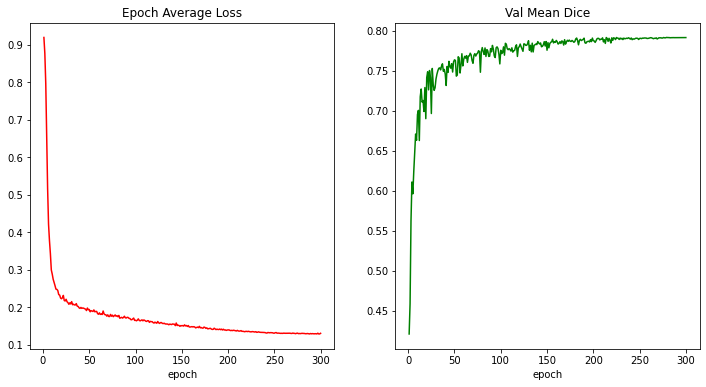
損失とメトリックのプロット
plt.figure("train", (12, 6))
plt.subplot(1, 2, 1)
plt.title("Epoch Average Loss")
x = [i + 1 for i in range(len(epoch_loss_values))]
y = epoch_loss_values
plt.xlabel("epoch")
plt.plot(x, y, color="red")
plt.subplot(1, 2, 2)
plt.title("Val Mean Dice")
x = [val_interval * (i + 1) for i in range(len(metric_values))]
y = metric_values
plt.xlabel("epoch")
plt.plot(x, y, color="green")
plt.show()
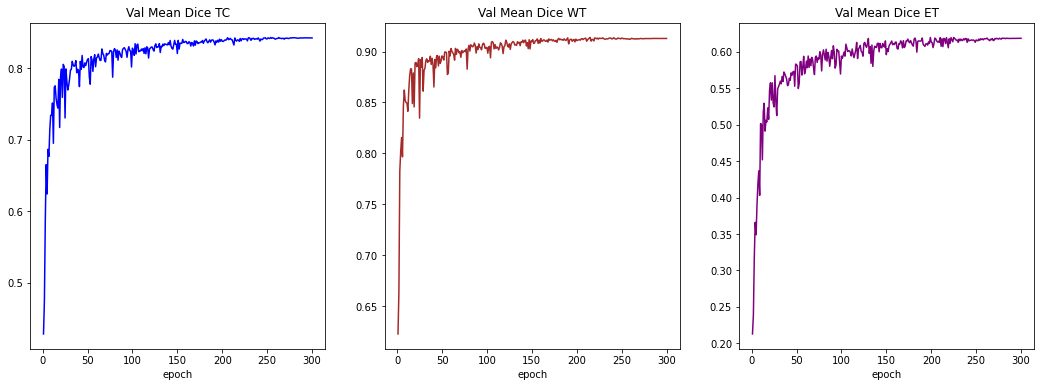
plt.figure("train", (18, 6))
plt.subplot(1, 3, 1)
plt.title("Val Mean Dice TC")
x = [val_interval * (i + 1) for i in range(len(metric_values_tc))]
y = metric_values_tc
plt.xlabel("epoch")
plt.plot(x, y, color="blue")
plt.subplot(1, 3, 2)
plt.title("Val Mean Dice WT")
x = [val_interval * (i + 1) for i in range(len(metric_values_wt))]
y = metric_values_wt
plt.xlabel("epoch")
plt.plot(x, y, color="brown")
plt.subplot(1, 3, 3)
plt.title("Val Mean Dice ET")
x = [val_interval * (i + 1) for i in range(len(metric_values_et))]
y = metric_values_et
plt.xlabel("epoch")
plt.plot(x, y, color="purple")
plt.show()
入力画像とラベルでベストモデル出力を確認する
model.load_state_dict(
torch.load(os.path.join(root_dir, "best_metric_model.pth"))
)
model.eval()
with torch.no_grad():
# select one image to evaluate and visualize the model output
val_input = val_ds[6]["image"].unsqueeze(0).to(device)
roi_size = (128, 128, 64)
sw_batch_size = 4
val_output = inference(val_input)
val_output = post_trans(val_output[0])
plt.figure("image", (24, 6))
for i in range(4):
plt.subplot(1, 4, i + 1)
plt.title(f"image channel {i}")
plt.imshow(val_ds[6]["image"][i, :, :, 70].detach().cpu(), cmap="gray")
plt.show()
# visualize the 3 channels label corresponding to this image
plt.figure("label", (18, 6))
for i in range(3):
plt.subplot(1, 3, i + 1)
plt.title(f"label channel {i}")
plt.imshow(val_ds[6]["label"][i, :, :, 70].detach().cpu())
plt.show()
# visualize the 3 channels model output corresponding to this image
plt.figure("output", (18, 6))
for i in range(3):
plt.subplot(1, 3, i + 1)
plt.title(f"output channel {i}")
plt.imshow(val_output[i, :, :, 70].detach().cpu())
plt.show()


元の画像 spacings 上の評価
val_org_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys=["image"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Orientationd(keys=["image"], axcodes="RAS"),
Spacingd(keys=["image"], pixdim=(1.0, 1.0, 1.0), mode="bilinear"),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
]
)
val_org_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=val_org_transforms,
section="validation",
download=False,
num_workers=4,
cache_num=0,
)
val_org_loader = DataLoader(val_org_ds, batch_size=1, shuffle=False, num_workers=4)
post_transforms = Compose([
Invertd(
keys="pred",
transform=val_org_transforms,
orig_keys="image",
meta_keys="pred_meta_dict",
orig_meta_keys="image_meta_dict",
meta_key_postfix="meta_dict",
nearest_interp=False,
to_tensor=True,
device="cpu",
),
Activationsd(keys="pred", sigmoid=True),
AsDiscreted(keys="pred", threshold=0.5),
])
model.load_state_dict(torch.load(
os.path.join(root_dir, "best_metric_model.pth")))
model.eval()
with torch.no_grad():
for val_data in val_org_loader:
val_inputs = val_data["image"].to(device)
val_data["pred"] = inference(val_inputs)
val_data = [post_transforms(i) for i in decollate_batch(val_data)]
val_outputs, val_labels = from_engine(["pred", "label"])(val_data)
dice_metric(y_pred=val_outputs, y=val_labels)
dice_metric_batch(y_pred=val_outputs, y=val_labels)
metric_org = dice_metric.aggregate().item()
metric_batch_org = dice_metric_batch.aggregate()
dice_metric.reset()
dice_metric_batch.reset()
metric_tc, metric_wt, metric_et = metric_batch_org[0].item(), metric_batch_org[1].item(), metric_batch_org[2].item()
print("Metric on original image spacing: ", metric_org)
print(f"metric_tc: {metric_tc:.4f}")
print(f"metric_wt: {metric_wt:.4f}")
print(f"metric_et: {metric_et:.4f}")
Metric on original image spacing: 0.7912478446960449 metric_tc: 0.8422 metric_wt: 0.9129 metric_et: 0.6187
データディレクトリのクリーンアップ
一時ディレクトリが使用された場合ディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
MONAI : What’s new in 1.1
MONAI : What’s new in 1.1 (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 01/17/2023
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP
MONAI : What’s new in 1.1
- デジタルパソロジー・ワークフロー
- MONAI バンドル用実験管理
- Auto3dSeg 拡張
- MONAI モデル Zoo の新しいモデル
- 最先端の SurgToolLoc ソリューション
デジタルパソロジー・ワークフロー
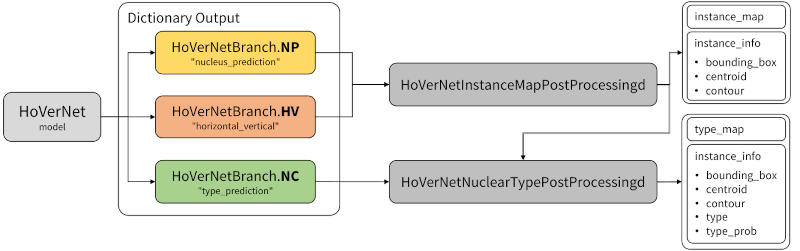
Hover は simultaneous segmentation and classification of nuclei in multi-tissue histology images (多組織の組織構造画像の核の同時に行われるセグメンテーションと分類) (Graham et al. Medical Image Analysis, 2019) のためのモデルです。私たちは、幾つかの新しいコンポーネントを実装し、既存のものを拡張し、そして訓練、検証と推論用のパイプラインとサンプルを提供することで MONAI でこのモデルのためのサポートを追加しました。
モジュールのリリースとともに、新しいデジタルパソロジー分析チュートリアルが利用可能になりました :
- 訓練、検証と推論用の MONAI ワークフローに基づく HoVerNet パイプライン
- 訓練、検証と推論用の HoVerNet チュートリアル
- 訓練 と 推論 用の NuClick (パソロジー用の対話的アノテーション) チュートリアル
- 訓練 と 推論 用の核分類チュートリアル
MONAI バンドル用実験管理
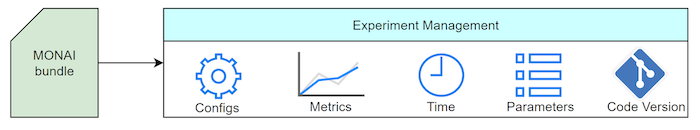
このリリースでは、実験管理機能が MONAI バンドルと統合されました。それは end-to-end なモデルバンドルのライフサイクルを管理するために必須な API を提供しています。例えば訓練 or 推論コマンドに MLFlow ベースの管理を有効にする –tracking “mlflow” を追加することで、ユーザは実験を追跡し始めることができます。デフォルトでは、MLFlow は実行されたバンドル config, モデル品質測定, そしてソースコードバージョン処理を追跡します。詳細は、チュートリアル を参照してください。
Auto3dSeg 拡張
ユーザビリティとパフォーマンスの両方の観点から Auto3DSeg に複数の改良が追加されました。
- HECKTOR22 チャレンジ・データセットの自動セグメンテーションのためにマルチ・モダリティのサポートが追加されて適用されました、これは様々な解像度とサイズの 3D CT と PET 画像の入力を含みます。HECKTOR22 チャレンジデータセット上で Auto3DSeg を実行するチュートリアルサンプルが MONAI チュートリアルで利用可能です。このチュートリアルは HECKTOR22 チャレンジ 1 位のソリューション に基づいています。
- Segresnet Algo の新しい改良版が AutoRunner で利用可能になりました。このバージョンでは、データキャッシュはより効率的で前処理変換はより柔軟です。ステップのタイミングも含めてワークフローの進捗は YAML ファイルに加えてコンソール出力に書かれます。
- モデル訓練 configuration の自動カスタマイゼーションと最適化は使用される GPU デバイスに従って得られます。この機能は、モデル訓練とスライディングウィンドウ推論のバッチサイズ、スライディングウィンドウ推論のデータに対する割り当てデバイス等のパラメータの決定にフォーカスしています。それを有効にする方法の詳細は、チュートリアル をご覧ください。
MONAI モデル Zoo の新しいモデル
新しい事前訓練済みモデルが作成されてモデル Zoo で公開されています。特に、
- mednist_reg は MONAI バンドル形式で画像レジストレーション・ワークフローを構築する方法を実演します。このモデルは registration_mednist チュートリアル に基づいて、手の X 線画像レジストレーションに対して ResNet と空間的 transformer を使用しています。
- pathology_nuclei_segmentation_and_classification, pathology_nuclick_annotation, そして pathology_nuclei_classification バンドルが デジタルパソロジー画像分析 のために構築されています。
モデルの使用方法の詳細は、チュートリアル をご覧ください。
最先端の SurgToolLoc ソリューション
SurgToolLoc は MICCAI 20222 の EndoVis チャレンジの一部です。チャレンジは内視鏡動画分析にフォーカスし、(1) 完全教師ありツール分類と (2) 弱教師ありツール分類/ローカリゼーションに分かれています。チーム NVIDIA は両方のカテゴリーを 3 位 で終えて入賞しました。ソリューションのコアコンポーネントは MONAI でリリース されています。
以上
MONAI : What’s new in 1.0
MONAI : What’s new in 1.0 (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 01/16/2023
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP
MONAI : What’s new in 1.0
- モデル Zoo
- Auto3DSeg
- 連合学習クライアント
- デジタルパソロジー・ワークフロー用の MetaTensor サポート
- 高速化 MRI 再構成
モデル Zoo
MONAI モデル Zoo は研究者やデータサイエンティストがコミュニティからの最新の素晴らしいモデルを使用し共有するための場所です。MONAI バンドル形式 の利用は任意の MONAI フレームワーク(Core、Label、Deploy)で任意のモデルを素早く使用し始めることを簡単にします。また、貴方のモデルを提供する ことに関心があれば、contributing ガイドラインをご覧ください、これはモデルを提供するためのプロセスと要件を案内します。モデルを使用する方法の詳細は、チュートリアル を見てください。
Auto3DSeg
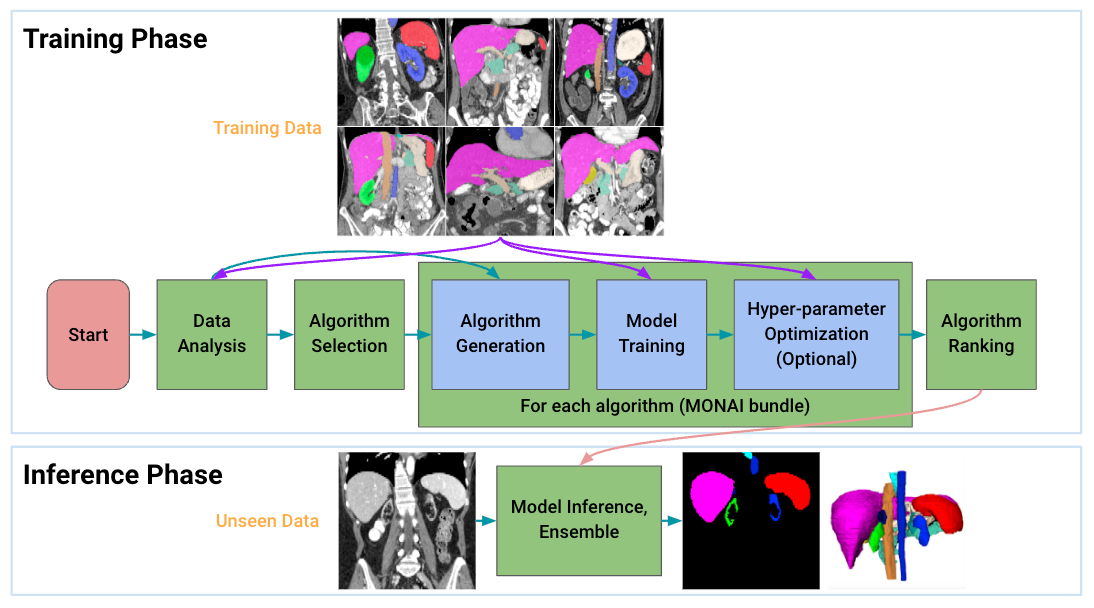
Auto3DSeg は大規模な 3D 医用画像セグメンテーションのための包括的なソリューションです。それは最先端の性能を持つアルゴリズムを効率的に開発して配備するために MONAI と GPU の最新の進歩を活用しています。それはデータセットの強度、次元性と解像度のようなグローバルな情報を最初に分析してから、データ統計と アルゴリズム・テンプレート に基づいて MONAI バンドル形式でアルゴリズムを生成します。次に、すべてのアルゴリズムはベストな検証性能を持つチェックポイントを得るためにモデル訓練を開始します。最後に、アンサンブル・モジュールが訓練済みチェックポイントのランク付けによりアルゴリズムを選択し、アンサンブル予測を作成します。
このソリューションは初心者から上級研究者に対して異なるレベルのユーザ経験を提供します。それは様々なモダリティの大規模 3D 医用画像データセットでテストされています。
連合学習クライアント
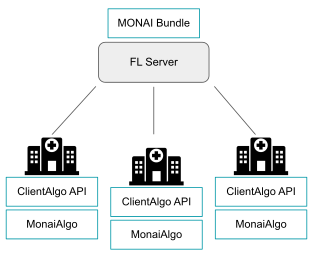
MONAI は連合学習 (FL) クライアント・アルゴリズム API を含むようになりました、これは任意の連合学習プラットフォームで実行されるアルゴリズムを定義するために抽象基底クラスとして公開されています。NVIDIA により開発された連合学習プラットフォーム、NVIDIA FLARE はこれらの新しい API を使用して 統合ピース を既に構築しています。新しい連合学習 API により、MONAI バンドルはシームレスに連合パラダイムに拡張され、シングル- or マルチ-GPU 訓練を使用して実行できます。MONAI FL クライアントはまたバンドル config で定義されたデータセット上でデータ統計の要約 (e.g., 強度ヒストグラム) を計算することを可能にします。これらは例えば NVIDIA FLARE の連合統計オペレータを使用して、FL サーバで共有して可視化することができます、サンプルは ここ をご覧ください。
私たちは他の連合学習ツールキットが MONAI FL API を使用して統合し、医用画像の collaborative (協調, 共同) 学習のための共通基盤を構築することを歓迎します。
デジタルパソロジー・ワークフロー用の MetaTensor サポート
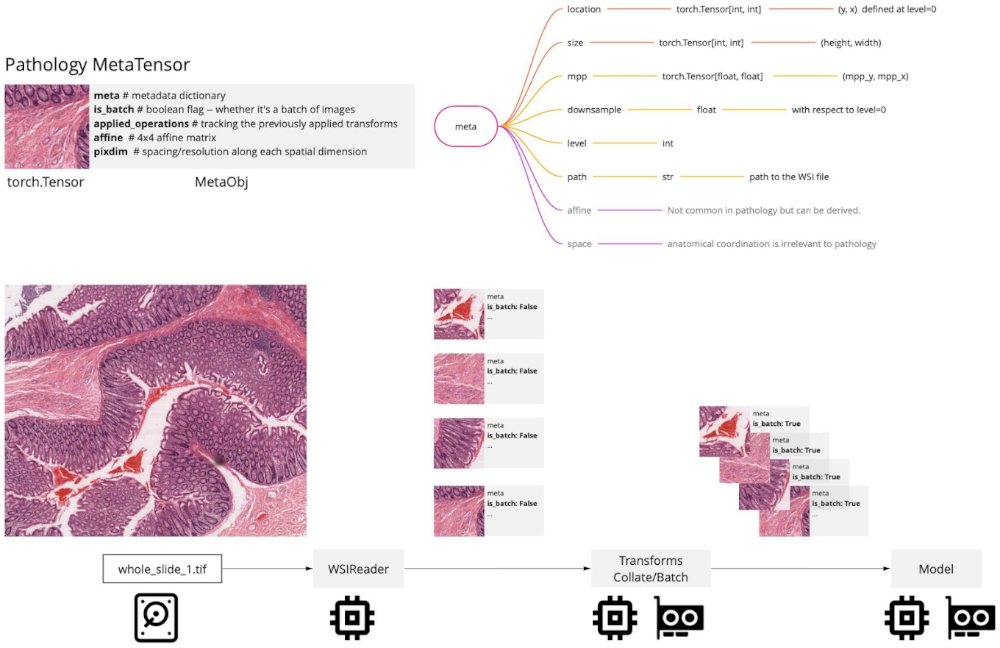
このリリースでは、すべてのデジタルパソロジー・コンポーネントで MetaTensor をサポートし、将来的な開発がそれらから恩恵を受けられることを確実にします。MONAI パソロジー・ワーキンググループの支援で、WSI から抽出された画像のパッチのメタデータ属性のセットを標準化して、再現性を保証して標準的な属性セットに依存することにより機能を拡張することができました。上図はすべてのパソロジーメタデータ属性とそれらの MetaTensors との関係性を示しています。チュートリアルとサンプル を見てください。
高速化 MRI 再構成
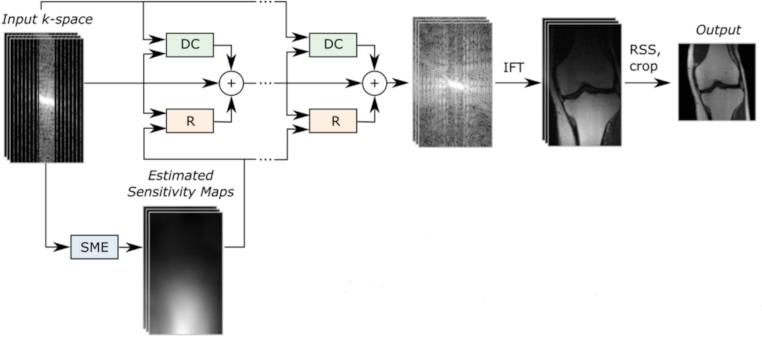
このリリースは様々なポピュラーな高速化 MRI 再構成ワークフローのために初期コンポーネントを含みます。それらの多くは汎用目的ツールで、例えば SSIMLoss 関数です。幾つかの新しい機能はタスク固有で、例えば FastMRIReader です。
詳細は、このタスクのためのベースラインモデルを使用するためには このチュートリアル を、最先端モデルを使用するためには このチュートリアル をご覧ください。
以上
MONAI 1.0 : tutorials : モジュール – GAN ワークフロー・エンジン (辞書版)
MONAI 1.0 : tutorials : モジュール – GAN ワークフロー・エンジン (辞書版) (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 11/13/2022 (1.0.1)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。
MONAI 1.0 : tutorials : モジュール – GAN ワークフロー・エンジン (辞書版)
このノートブックは GanTrainer、モジュール化された敵対的学習のための MONAI ワークフロー・エンジンを紹介します。MedNIST ハンド CT スキャン・データセットを使用して医用画像再構築ネットワークを訓練します。辞書バージョンです。
MONAI フレームワークは敵対的生成ネットワークを簡単に設計し、訓練して評価するために使用できます。このノートブックは、ハンド CT スキャンの画像を再構築するために単純な GAN モデルを設計して訓練する MONAI コンポーネントを使用する実例を示します。
ネットワーク・アーキテクチャと損失関数の詳細は MONAI Mednist GAN チュートリアル を読んでください。
Step 1: セットアップ
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[ignite, tqdm]"
!python -c "import matplotlib" || pip install -q matplotlib
%matplotlib inline
from monai.utils import set_determinism
from monai.transforms import (
AddChannelD,
Compose,
LoadImageD,
RandFlipD,
RandRotateD,
RandZoomD,
ScaleIntensityD,
EnsureTypeD,
)
from monai.networks.nets import Discriminator, Generator
from monai.networks import normal_init
from monai.handlers import CheckpointSaver, MetricLogger, StatsHandler
from monai.engines.utils import GanKeys, default_make_latent
from monai.engines import GanTrainer
from monai.data import CacheDataset, DataLoader
from monai.config import print_config
from monai.apps import download_and_extract
import numpy as np
import torch
import matplotlib.pyplot as plt
import tempfile
import sys
import shutil
import os
import logging
インポートのセットアップ
print_config()
MONAI version: 0.6.0rc1+23.gc6793fd0
Numpy version: 1.20.3
Pytorch version: 1.9.0a0+c3d40fd
MONAI flags: HAS_EXT = True, USE_COMPILED = False
MONAI rev id: c6793fd0f316a448778d0047664aaf8c1895fe1c
Optional dependencies:
Pytorch Ignite version: 0.4.5
Nibabel version: 3.2.1
scikit-image version: 0.15.0
Pillow version: 7.0.0
Tensorboard version: 2.5.0
gdown version: 3.13.0
TorchVision version: 0.10.0a0
ITK version: 5.1.2
tqdm version: 4.53.0
lmdb version: 1.2.1
psutil version: 5.8.0
pandas version: 1.1.4
einops version: 0.3.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。
これは結果をセーブしてダウンロードを再利用することを可能にします。
指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical
データセットをダウンロードする
データセットをダウンロードして展開します。
MedMNIST データセットは TCIA, RSNA Bone Age チャレンジ と NIH Chest X-ray データセット からの様々なセットから集められました。
データセットは Dr. Bradley J. Erickson M.D., Ph.D. (Department of Radiology, Mayo Clinic) のお陰により Creative Commons CC BY-SA 4.0 ライセンス のもとで利用可能になっています。
MedNIST データセットを使用する場合、出典を明示してください、e.g. https://github.com/Project-MONAI/tutorials/blob/master/2d_classification/mednist_tutorial.ipynb。
resource = "https://github.com/Project-MONAI/MONAI-extra-test-data/releases/download/0.8.1/MedNIST.tar.gz"
md5 = "0bc7306e7427e00ad1c5526a6677552d"
compressed_file = os.path.join(root_dir, "MedNIST.tar.gz")
data_dir = os.path.join(root_dir, "MedNIST")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
hand_dir = os.path.join(data_dir, "Hand")
training_datadict = [
{"hand": os.path.join(hand_dir, filename)}
for filename in os.listdir(hand_dir)
]
print(training_datadict[:5])
[{'hand': '/workspace/data/medical/MedNIST/Hand/000317.jpeg'}, {'hand': '/workspace/data/medical/MedNIST/Hand/002344.jpeg'}, {'hand': '/workspace/data/medical/MedNIST/Hand/000816.jpeg'}, {'hand': '/workspace/data/medical/MedNIST/Hand/004046.jpeg'}, {'hand': '/workspace/data/medical/MedNIST/Hand/003316.jpeg'}]
Step 2: MONAI コンポーネントを初期化する
logging.basicConfig(stream=sys.stdout, level=logging.INFO)
set_determinism(0)
device = torch.device("cuda:0")
画像変換チェインを作成する
セーブされたディスク画像を利用可能なテンソルに変換するために処理パイプラインを定義します。
train_transforms = Compose(
[
LoadImageD(keys=["hand"]),
AddChannelD(keys=["hand"]),
ScaleIntensityD(keys=["hand"]),
RandRotateD(keys=["hand"], range_x=np.pi /
12, prob=0.5, keep_size=True),
RandFlipD(keys=["hand"], spatial_axis=0, prob=0.5),
RandZoomD(keys=["hand"], min_zoom=0.9, max_zoom=1.1, prob=0.5),
EnsureTypeD(keys=["hand"]),
]
)
データセットとデータローダを作成する
データを保持して訓練の間にバッチを提示します。
real_dataset = CacheDataset(training_datadict, train_transforms)
100%|██████████| 10000/10000 [00:09<00:00, 1000.72it/s]
batch_size = 300
real_dataloader = DataLoader(
real_dataset, batch_size=batch_size, shuffle=True, num_workers=10)
def prepare_batch(batchdata, device=None, non_blocking=False):
return batchdata["hand"].to(device=device, non_blocking=non_blocking)
generator と discriminator を定義する
基本的なコンピュータビジョン GAN ネットワークをライブラリからロードします。
# define networks
disc_net = Discriminator(
in_shape=(1, 64, 64),
channels=(8, 16, 32, 64, 1),
strides=(2, 2, 2, 2, 1),
num_res_units=1,
kernel_size=5,
).to(device)
latent_size = 64
gen_net = Generator(
latent_shape=latent_size,
start_shape=(latent_size, 8, 8),
channels=[32, 16, 8, 1],
strides=[2, 2, 2, 1],
)
gen_net.conv.add_module("activation", torch.nn.Sigmoid())
gen_net = gen_net.to(device)
# initialize both networks
disc_net.apply(normal_init)
gen_net.apply(normal_init)
# define optimizors
learning_rate = 2e-4
betas = (0.5, 0.999)
disc_opt = torch.optim.Adam(disc_net.parameters(), learning_rate, betas=betas)
gen_opt = torch.optim.Adam(gen_net.parameters(), learning_rate, betas=betas)
# define loss functions
disc_loss_criterion = torch.nn.BCELoss()
gen_loss_criterion = torch.nn.BCELoss()
real_label = 1
fake_label = 0
def discriminator_loss(gen_images, real_images):
real = real_images.new_full((real_images.shape[0], 1), real_label)
gen = gen_images.new_full((gen_images.shape[0], 1), fake_label)
realloss = disc_loss_criterion(disc_net(real_images), real)
genloss = disc_loss_criterion(disc_net(gen_images.detach()), gen)
return (genloss + realloss) / 2
def generator_loss(gen_images):
output = disc_net(gen_images)
cats = output.new_full(output.shape, real_label)
return gen_loss_criterion(output, cats)
訓練ハンドラを作成する
モデル訓練の間に操作を実行します。
metric_logger = MetricLogger(
loss_transform=lambda x: {
GanKeys.GLOSS: x[GanKeys.GLOSS], GanKeys.DLOSS: x[GanKeys.DLOSS]},
metric_transform=lambda x: x,
)
handlers = [
StatsHandler(
name="batch_training_loss",
output_transform=lambda x: {
GanKeys.GLOSS: x[GanKeys.GLOSS],
GanKeys.DLOSS: x[GanKeys.DLOSS],
},
),
CheckpointSaver(
save_dir=os.path.join(root_dir, "hand-gan"),
save_dict={"g_net": gen_net, "d_net": disc_net},
save_interval=10,
save_final=True,
epoch_level=True,
),
metric_logger,
]
GanTrainer を作成する
敵対的学習のための MONAI ワークフロー・エンジン。GanTrainer によってコンポーネントはここで集められます。
Goodfellow et al. 2014 https://arxiv.org/abs/1406.2661 に基づいた訓練ループを使用します。
- ランダムな潜在コードから m 個の fakes を生成します。
- これらの fakes と現在のバッチ reals で D を更新します、d_train_steps 回反復されます。
- 新しいランダムな潜在コードから m fakes を生成します。
- discriminator フィードバックを使用してこれらの fakes で generator を更新します。
disc_train_steps = 5
max_epochs = 50
trainer = GanTrainer(
device,
max_epochs,
real_dataloader,
gen_net,
gen_opt,
generator_loss,
disc_net,
disc_opt,
discriminator_loss,
d_prepare_batch=prepare_batch,
d_train_steps=disc_train_steps,
g_update_latents=True,
latent_shape=latent_size,
key_train_metric=None,
train_handlers=handlers,
)
Step 3: 訓練の開始
trainer.run()
結果を評価する
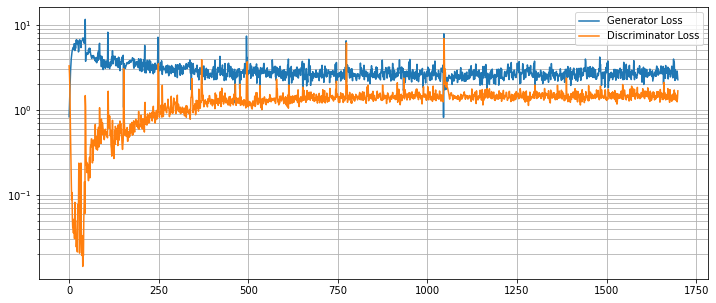
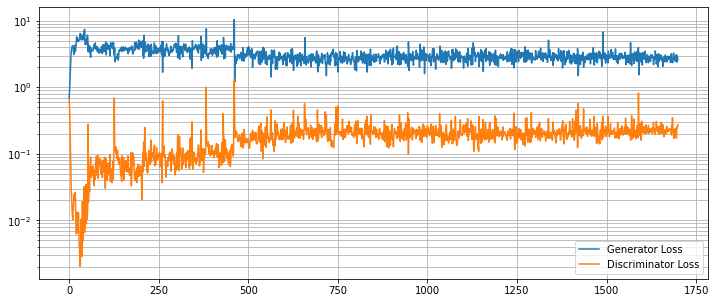
G と D の損失カーブを崩れていないか調べます。
g_loss = [loss[1][GanKeys.GLOSS] for loss in metric_logger.loss]
d_loss = [loss[1][GanKeys.DLOSS] for loss in metric_logger.loss]
plt.figure(figsize=(12, 5))
plt.semilogy(g_loss, label="Generator Loss")
plt.semilogy(d_loss, label="Discriminator Loss")
plt.grid(True, "both", "both")
plt.legend()
plt.show()
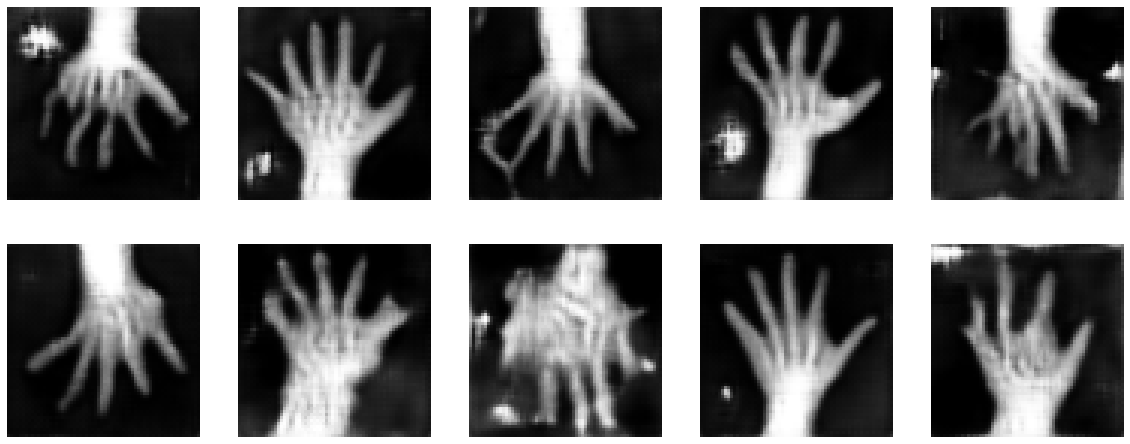
画像再構築を見る
ランダムな潜在コードで訓練された generator の出力を見ます。
test_img_count = 10
test_latents = default_make_latent(test_img_count, latent_size).to(device)
fakes = gen_net(test_latents)
fig, axs = plt.subplots(2, (test_img_count // 2), figsize=(20, 8))
axs = axs.flatten()
for i, ax in enumerate(axs):
ax.axis("off")
ax.imshow(fakes[i, 0].cpu().data.numpy(), cmap="gray")

データディレクトリのクリーンアップ
一時ディレクトリが作成された場合にはディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
MONAI 1.0 : tutorials : モジュール – 2D 画像変換デモ
MONAI 1.0 : tutorials : モジュール – 2D 画像変換デモ (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 11/11/2022 (1.0.1)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。
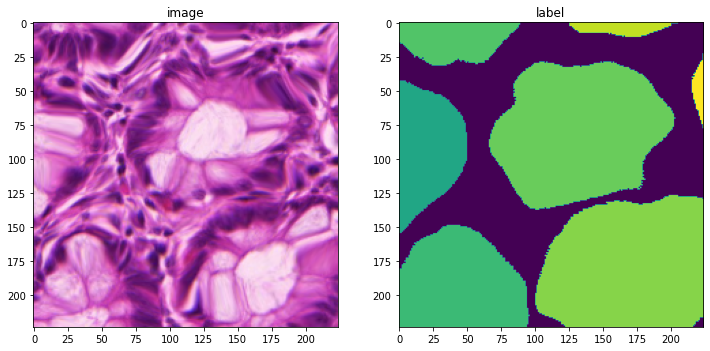
MONAI 1.0 : tutorials : モジュール – 2D 画像変換デモ
このノートブックは GlaS Contest データセット を使用して組織学 (= histology) の画像の画像変換を実演します。
このデモは MONAI の 2D 変換を適用する方法を示します。主な特徴は :
- ネイティブ PyTorch で実装されたランダムな elastic 変換
- pythonic な方法で設計されて実装された使いやすいインターフェイス
詳細は MONAI の wiki ページで調べてください : https://github.com/Project-MONAI/MONAI/wiki
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[pillow,tqdm]"
!python -c "import matplotlib" || pip install -q matplotlib
%matplotlib inline
from monai.transforms import Affine, Rand2DElastic
from monai.config import print_config
from monai.apps import download_and_extract
import torch
import PIL
import numpy as np
import matplotlib.pyplot as plt
import tempfile
import shutil
import os
インポートのセットアップ
# Copyright 2020 MONAI Consortium
# Licensed under the Apache License, Version 2.0 (the "License");
# you may not use this file except in compliance with the License.
# You may obtain a copy of the License at
# http://www.apache.org/licenses/LICENSE-2.0
# Unless required by applicable law or agreed to in writing, software
# distributed under the License is distributed on an "AS IS" BASIS,
# WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied.
# See the License for the specific language governing permissions and
# limitations under the License.
print_config()
MONAI version: 0.6.0+135.g3cb355bb
Numpy version: 1.21.2
Pytorch version: 1.9.0
MONAI flags: HAS_EXT = False, USE_COMPILED = False
MONAI rev id: 3cb355bb4e50702a17854cea1b817076c021c1b5
Optional dependencies:
Pytorch Ignite version: 0.4.5
Nibabel version: 3.2.1
scikit-image version: 0.18.3
Pillow version: 8.3.2
Tensorboard version: 2.6.0
gdown version: 3.13.0
TorchVision version: 0.10.0
tqdm version: 4.62.2
lmdb version: 1.2.1
psutil version: 5.8.0
pandas version: 1.3.3
einops version: 0.3.2
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。
これは結果をセーブしてダウンロードを再利用することを可能にします。
指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/var/folders/6f/fdkl7m0x7sz3nj_t7p3ccgz00000gp/T/tmpo87g5vor
データセットのダウンロード
データセットをダウンロードして解凍します。データセットは https://warwick.ac.uk/fac/sci/dcs/research/tia/glascontest/ に由来します。
このコンペで使用されたデータセットは研究目的のみで提供されます。商用利用は許容されません。
K. Sirinukunwattana, J. P. W. Pluim, H. Chen, X Qi, P. Heng, Y. Guo, L. Wang, B. J. Matuszewski, E. Bruni, U. Sanchez, A. Böhm, O. Ronneberger, B. Ben Cheikh, D. Racoceanu, P. Kainz, M. Pfeiffer, M. Urschler, D. R. J. Snead, N. M. Rajpoot, “Gland Segmentation in Colon Histology Images: The GlaS Challenge Contest” http://arxiv.org/abs/1603.00275
K. Sirinukunwattana, D.R.J. Snead, N.M. Rajpoot, “A Stochastic Polygons Model for Glandular Structures in Colon Histology Images,” in IEEE Transactions on Medical Imaging, 2015 doi: 10.1109/TMI.2015.2433900
resource = "https://warwick.ac.uk/fac/sci/dcs/research/tia/" \
+ "glascontest/download/warwick_qu_dataset_released_2016_07_08.zip"
md5 = None
compressed_file = os.path.join(
root_dir, "warwick_qu_dataset_released_2016_07_08.zip")
data_dir = os.path.join(root_dir, "Warwick QU Dataset (Released 2016_07_08)")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
warwick_qu_dataset_released_2016_07_08.zip: 173MB [00:20, 8.95MB/s]
Downloaded: /var/folders/6f/fdkl7m0x7sz3nj_t7p3ccgz00000gp/T/tmpo87g5vor/warwick_qu_dataset_released_2016_07_08.zip Expected md5 is None, skip md5 check for file /var/folders/6f/fdkl7m0x7sz3nj_t7p3ccgz00000gp/T/tmpo87g5vor/warwick_qu_dataset_released_2016_07_08.zip. Writing into directory: /var/folders/6f/fdkl7m0x7sz3nj_t7p3ccgz00000gp/T/tmpo87g5vor.
device = "cpu" if not torch.cuda.is_available() else "cuda:0"
img_name = os.path.join(data_dir, "train_22.bmp")
seg_name = os.path.join(data_dir, "train_22_anno.bmp")
im = np.array(PIL.Image.open(img_name))
seg = np.array(PIL.Image.open(seg_name))
plt.figure("check", (12, 6))
plt.subplot(1, 2, 1)
plt.title("image")
plt.imshow(im)
plt.subplot(1, 2, 2)
plt.title("label")
plt.imshow(seg)
plt.show()
print(im.shape, seg.shape)
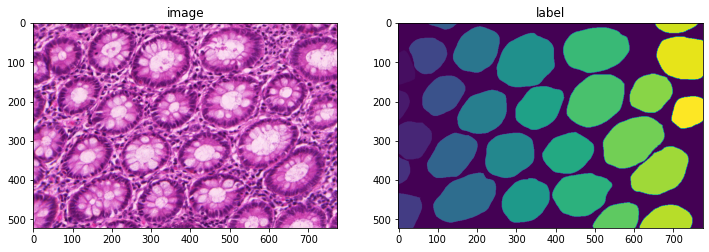
(522, 775, 3) (522, 775)
アフィン変換
# MONAI transforms always take channel-first data: [channel x H x W]
im_data = np.moveaxis(im, -1, 0) # make them channel first
seg_data = np.expand_dims(seg, 0) # make a channel for the segmentation
# create an Affine transform
affine = Affine(
rotate_params=np.pi / 4,
scale_params=(1.2, 1.2),
translate_params=(200, 40),
padding_mode="zeros",
device=device,
)
# convert both image and segmentation using different interpolation mode
new_img, _ = affine(im_data, (300, 400), mode="bilinear")
new_seg, _ = affine(seg_data, (300, 400), mode="nearest")
print(new_img.shape, new_seg.shape)
(3, 300, 400) (1, 300, 400)
plt.figure("check", (12, 6))
plt.subplot(1, 2, 1)
plt.title("image")
plt.imshow(np.moveaxis(new_img.astype(int), 0, -1))
plt.subplot(1, 2, 2)
plt.title("label")
plt.imshow(new_seg[0].astype(int))
plt.show()
Elastic 変形 (= deformation)
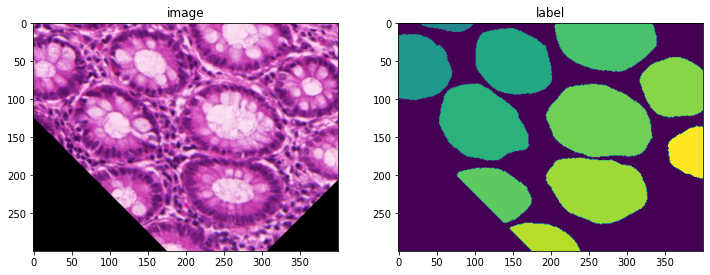
# create an elsatic deformation transform
deform = Rand2DElastic(
prob=1.0,
spacing=(30, 30),
magnitude_range=(5, 6),
rotate_range=(np.pi / 4,),
scale_range=(0.2, 0.2),
translate_range=(100, 100),
padding_mode="zeros",
device=device,
)
# transform both image and segmentation using different interpolation mode
deform.set_random_state(seed=123)
new_img = deform(im_data, (224, 224), mode="bilinear")
deform.set_random_state(seed=123)
new_seg = deform(seg_data, (224, 224), mode="nearest")
print(new_img.shape, new_seg.shape)
(3, 224, 224) (1, 224, 224)
plt.figure("check", (12, 6))
plt.subplot(1, 2, 1)
plt.title("image")
plt.imshow(np.moveaxis(new_img.astype(int), 0, -1))
plt.subplot(1, 2, 2)
plt.title("label")
plt.imshow(new_seg[0].astype(int))
plt.show()

データディレクトリのクリーンアップ
一時ディレクトリが使用された場合ディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
MONAI 1.0 : tutorials : モジュール – MedNIST で GAN
MONAI 1.0 : tutorials : モジュール – MedNIST で GAN (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 11/08/2021 (1.0.1)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP
MONAI 1.0 : tutorials : モジュール – MedNIST で GAN
このノートブックはランダムな入力テンソルから画像を生成するネットワークを訓練するための MONAI の使用方法を例示します。個別の Generator と Discriminator ネットワークに対処する単純な GAN が採用されます。
これは以下のステップを進みます :
- 遠隔ソースからデータをロードする
- このデータからのデータセットと変換を構築する
- ネットワークを定義する
- 訓練と評価
環境セットアップ
!python -c "import monai" || pip install -q monai-weekly
!python -c "import matplotlib" || pip install -q matplotlib
%matplotlib inline
from monai.utils import progress_bar, set_determinism
from monai.transforms import (
AddChannel,
Compose,
RandFlip,
RandRotate,
RandZoom,
ScaleIntensity,
EnsureType,
Transform,
)
from monai.networks.nets import Discriminator, Generator
from monai.networks import normal_init
from monai.data import CacheDataset
from monai.config import print_config
from monai.apps import download_and_extract
import numpy as np
import torch
import matplotlib.pyplot as plt
import os
import tempfile
インポートのセットアップ
# Copyright 2020 MONAI Consortium
# Licensed under the Apache License, Version 2.0 (the "License");
# you may not use this file except in compliance with the License.
# You may obtain a copy of the License at
# http://www.apache.org/licenses/LICENSE-2.0
# Unless required by applicable law or agreed to in writing, software
# distributed under the License is distributed on an "AS IS" BASIS,
# WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied.
# See the License for the specific language governing permissions and
# limitations under the License.
print_config()
MONAI version: 0.6.0rc1+23.gc6793fd0
Numpy version: 1.20.3
Pytorch version: 1.9.0a0+c3d40fd
MONAI flags: HAS_EXT = True, USE_COMPILED = False
MONAI rev id: c6793fd0f316a448778d0047664aaf8c1895fe1c
Optional dependencies:
Pytorch Ignite version: 0.4.5
Nibabel version: 3.2.1
scikit-image version: 0.15.0
Pillow version: 7.0.0
Tensorboard version: 2.5.0
gdown version: 3.13.0
TorchVision version: 0.10.0a0
ITK version: 5.1.2
tqdm version: 4.53.0
lmdb version: 1.2.1
psutil version: 5.8.0
pandas version: 1.1.4
einops version: 0.3.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
再現性のための決定論的訓練
set_determinism(seed=0)
訓練変数の定義
disc_train_interval = 1
disc_train_steps = 5
batch_size = 300
latent_size = 64
max_epochs = 50
real_label = 1
gen_label = 0
learning_rate = 2e-4
betas = (0.5, 0.999)
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。
これは結果をセーブしてダウンロードを再利用することを可能にします。
指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical
データセットをダウンロードする
MedMNIST データセットは TCIA, RSNA Bone Age チャレンジ と NIH Chest X-ray データセット からの様々なセットから集められました。
データセットは Dr. Bradley J. Erickson M.D., Ph.D. (Department of Radiology, Mayo Clinic) のお陰により Creative Commons CC BY-SA 4.0 ライセンス のもとで利用可能になっています。
MedNIST データセットを使用する場合、出典を明示してください、e.g. https://github.com/Project-MONAI/tutorials/blob/master/2d_classification/mednist_tutorial.ipynb。
ここではファイルシステムを使用することなく tar ファイルからダウンロードして読む方法を示すためと、ハンド X-rays の画像だけを望むために、遠隔ソースからデータをロードする方法は異なります。これは分類サンプルではないのでカテゴリーデータは必要ありませんので、tarball をダウンロードし、標準ライブラリを使用してそれをオープンし、そしてハンドのためのファイル名の総てを recall します :
resource = "https://github.com/Project-MONAI/MONAI-extra-test-data/releases/download/0.8.1/MedNIST.tar.gz"
md5 = "0bc7306e7427e00ad1c5526a6677552d"
compressed_file = os.path.join(root_dir, "MedNIST.tar.gz")
data_dir = os.path.join(root_dir, "MedNIST")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
hands = [
os.path.join(data_dir, "Hand", x)
for x in os.listdir(os.path.join(data_dir, "Hand"))
]
tarfile から実際の画像データをロードするため、Matplotlib を使用してこれを行なう変換タイプを定義します。これはデータを準備するために他の変換とともに使用され、ランダム化された増強変換が続きます。ここでは tarball からの準備された画像の総てをキャッシュするために CacheDataset クラスが使用されますので、ランダム化された回転、反転とズーム操作により増強されることになる準備された画像の総てをメモリに持ちます :
class LoadTarJpeg(Transform):
def __call__(self, data):
return plt.imread(data)
train_transforms = Compose(
[
LoadTarJpeg(),
AddChannel(),
ScaleIntensity(),
RandRotate(range_x=np.pi / 12, prob=0.5, keep_size=True),
RandFlip(spatial_axis=0, prob=0.5),
RandZoom(min_zoom=0.9, max_zoom=1.1, prob=0.5),
EnsureType(),
]
)
train_ds = CacheDataset(hands, train_transforms)
train_loader = torch.utils.data.DataLoader(
train_ds, batch_size=batch_size, shuffle=True, num_workers=10
)
100%|██████████| 10000/10000 [00:05<00:00, 1691.00it/s]
今は generator と discriminator ネットワークを定義します。パラメータは tar ファイルからロードされた (1, 64, 64) の画像サイズに合うように注意深く選択されています。discriminator への入力画像は非常に小さい画像を生成するために 4 回ダウンサンプリングされます、これらは平坦化されて完全結合層への入力として渡されます。generator への入力潜在ベクトルは shape (64, 8, 8) の出力を生成するために完全結合層に渡されます、そしてこれはリアル画像と同じ shape である最終的な出力を生成するために 3 回アップサンプリングされます。結果を改善するためにネットワークは正規化スキームで初期化されます :
device = torch.device("cuda:0")
disc_net = Discriminator(
in_shape=(1, 64, 64),
channels=(8, 16, 32, 64, 1),
strides=(2, 2, 2, 2, 1),
num_res_units=1,
kernel_size=5,
).to(device)
gen_net = Generator(
latent_shape=latent_size, start_shape=(64, 8, 8),
channels=[32, 16, 8, 1], strides=[2, 2, 2, 1],
)
# initialize both networks
disc_net.apply(normal_init)
gen_net.apply(normal_init)
# input images are scaled to [0,1] so enforce the same of generated outputs
gen_net.conv.add_module("activation", torch.nn.Sigmoid())
gen_net = gen_net.to(device)
今は generator と discriminator のための損失計算プロセスをラップするヘルパー関数とともに使用する損失関数を定義します。optimizer もまた定義します :
disc_loss = torch.nn.BCELoss()
gen_loss = torch.nn.BCELoss()
disc_opt = torch.optim.Adam(disc_net.parameters(), learning_rate, betas=betas)
gen_opt = torch.optim.Adam(gen_net.parameters(), learning_rate, betas=betas)
def discriminator_loss(gen_images, real_images):
"""
The discriminator loss if calculated by comparing its
prediction for real and generated images.
"""
real = real_images.new_full((real_images.shape[0], 1), real_label)
gen = gen_images.new_full((gen_images.shape[0], 1), gen_label)
realloss = disc_loss(disc_net(real_images), real)
genloss = disc_loss(disc_net(gen_images.detach()), gen)
return (realloss + genloss) / 2
def generator_loss(input):
"""
The generator loss is calculated by determining how well
the discriminator was fooled by the generated images.
"""
output = disc_net(input)
cats = output.new_full(output.shape, real_label)
return gen_loss(output, cats)
今は幾つかのエポックの間データセットに渡り反復することにより訓練します。各バッチのための generator 訓練ステージの後、discriminator は同じリアルと生成画像上で幾つかのステップの間訓練されます。
epoch_loss_values = [(0, 0)]
gen_step_loss = []
disc_step_loss = []
step = 0
for epoch in range(max_epochs):
gen_net.train()
disc_net.train()
epoch_loss = 0
for i, batch_data in enumerate(train_loader):
progress_bar(
i, len(
train_loader),
f"epoch {epoch + 1}, avg loss: {epoch_loss_values[-1][1]:.4f}",
)
real_images = batch_data.to(device)
latent = torch.randn(real_images.shape[0], latent_size).to(device)
gen_opt.zero_grad()
gen_images = gen_net(latent)
loss = generator_loss(gen_images)
loss.backward()
gen_opt.step()
epoch_loss += loss.item()
gen_step_loss.append((step, loss.item()))
if step % disc_train_interval == 0:
disc_total_loss = 0
for _ in range(disc_train_steps):
disc_opt.zero_grad()
dloss = discriminator_loss(gen_images, real_images)
dloss.backward()
disc_opt.step()
disc_total_loss += dloss.item()
disc_step_loss.append((step, disc_total_loss / disc_train_steps))
step += 1
epoch_loss /= step
epoch_loss_values.append((step, epoch_loss))
33/34 epoch 50, avg loss: 0.0563 [============================= ]
generator と discriminator のための個別の損失値は一緒にグラフ化できます。これらは、discriminator を騙す generator の能力がリアルとフェイク画像の間を正確に識別するネットワークの能力と均衡するにつれて、均衡に達するはずです。
plt.figure(figsize=(12, 5))
plt.semilogy(*zip(*gen_step_loss), label="Generator Loss")
plt.semilogy(*zip(*disc_step_loss), label="Discriminator Loss")
plt.grid(True, "both", "both")
plt.legend()

最後に幾つかランダムに生成された画像を示します。望ましくは期待されるように殆どの画像が 4 本の指と親指を持つことです (polydactyl (= 多指の) サンプルがデータセットに多くは存在していないと仮定して)。このデモ目的のノートブックは長くはネットワークを訓練しません、デフォルトの 50 エポックを越えた訓練は結果を改善するはずです。
test_size = 10
test_latent = torch.randn(test_size, latent_size).to(device)
test_images = gen_net(test_latent)
fig, axs = plt.subplots(1, test_size, figsize=(20, 4))
for i, ax in enumerate(axs):
ax.axis("off")
ax.imshow(test_images[i, 0].cpu().data.numpy(), cmap="gray")

以上
MONAI 1.0 : tutorials : 2D レジストレーション – 2D XRay レジストレーション・デモ
MONAI 1.0 : tutorials : 2D レジストレーション – 2D XRay レジストレーション・デモ (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 11/08/2021 (1.0.1)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP

MONAI 1.0 : tutorials : 2D レジストレーション – 2D XRay レジストレーション・デモ
このノートブックは学習ベースの 64 x 64 X-Ray ハンドのアフィン・レジストレーションの素早いデモを示します。
このデモは MONAI のレジストレーション機能の使用方法を示す toy サンプルです。
このデモは主として以下を使用します :
- アフィン変換パラメータを予測するためにアフィンヘッドを持つ UNet ライクなレジストレーション・ネットワーク ;
- moving 画像を変換するための、MONAI C++/CUDA として実装された、warp 関数。
環境のセットアップ
BUILD_MONAI=1 フラグで “pip install” すると、MONA レポジトリから最新のソースコードを取得し、MONAI の C++/CUDA 拡張を構築して、パッケージをインストールします。
env BUILD_MONAI=1 の設定は、関連する Python モジュールを呼び出すとき MONAI は Pytorch/Python ネイティブ実装の代わりにそれらの拡張を優先することを示します。
(コンパイルは数分から 10+ 分かかる場合があります。)
%env BUILD_MONAI=1
!python -c "import monai" || pip install -q git+https://github.com/Project-MONAI/MONAI#egg=monai[all]
インポートのセットアップ
from monai.utils import set_determinism, first
from monai.transforms import (
EnsureChannelFirstD,
Compose,
LoadImageD,
RandRotateD,
RandZoomD,
ScaleIntensityRanged,
)
from monai.data import DataLoader, Dataset, CacheDataset
from monai.config import print_config, USE_COMPILED
from monai.networks.nets import GlobalNet
from monai.networks.blocks import Warp
from monai.apps import MedNISTDataset
import numpy as np
import torch
from torch.nn import MSELoss
import matplotlib.pyplot as plt
import os
import tempfile
print_config()
set_determinism(42)
MONAI version: 0.5.0+7.g9f4da6a
Numpy version: 1.19.5
Pytorch version: 1.8.1+cu101
MONAI flags: HAS_EXT = True, USE_COMPILED = True
MONAI rev id: 9f4da6acded249bba24c85eaee4ece256ed45815
Optional dependencies:
Pytorch Ignite version: 0.4.4
Nibabel version: 3.0.2
scikit-image version: 0.16.2
Pillow version: 7.1.2
Tensorboard version: 2.4.1
gdown version: 3.6.4
TorchVision version: 0.9.1+cu101
ITK version: 5.1.2
tqdm version: 4.60.0
lmdb version: 0.99
psutil version: 5.4.8
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。
これは結果をセーブしてダウンロードを再利用することを可能にします。
指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
ペア単位の訓練入力の構築
実際のデータファイルをダウンロードして unzip するために MedNISTDataset オブジェクトを使用します。そして hand クラスを選択し、ロードされたデータ辞書を “fixed_hand” と “moving_hand” に変換します、これらは合成訓練ペアを作成するために別々に前処理されます。
train_data = MedNISTDataset(root_dir=root_dir, section="training", download=True, transform=None)
training_datadict = [
{"fixed_hand": item["image"], "moving_hand": item["image"]}
for item in train_data.data if item["label"] == 4 # label 4 is for xray hands
]
print("\n first training items: ", training_datadict[:3])
MedNIST.tar.gz: 59.0MB [00:07, 8.83MB/s]
downloaded file: ./MedNIST.tar.gz.
Verified 'MedNIST.tar.gz', md5: 0bc7306e7427e00ad1c5526a6677552d.
Verified 'MedNIST.tar.gz', md5: 0bc7306e7427e00ad1c5526a6677552d.
Loading dataset: 100%|██████████| 47164/47164 [00:00<00:00, 145309.19it/s]
first training items: [{'fixed_hand': './MedNIST/Hand/003696.jpeg', 'moving_hand': './MedNIST/Hand/003696.jpeg'}, {'fixed_hand': './MedNIST/Hand/001404.jpeg', 'moving_hand': './MedNIST/Hand/001404.jpeg'}, {'fixed_hand': './MedNIST/Hand/008882.jpeg', 'moving_hand': './MedNIST/Hand/008882.jpeg'}]
train_transforms = Compose(
[
LoadImageD(keys=["fixed_hand", "moving_hand"]),
EnsureChannelFirstD(keys=["fixed_hand", "moving_hand"]),
ScaleIntensityRanged(keys=["fixed_hand", "moving_hand"],
a_min=0., a_max=255., b_min=0.0, b_max=1.0, clip=True,),
RandRotateD(keys=["moving_hand"], range_x=np.pi/4, prob=1.0, keep_size=True, mode="bicubic"),
RandZoomD(keys=["moving_hand"], min_zoom=0.9, max_zoom=1.1, prob=1.0, mode="bicubic", align_corners=False),
]
)
訓練ペアの可視化
check_ds = Dataset(data=training_datadict, transform=train_transforms)
check_loader = DataLoader(check_ds, batch_size=1, shuffle=True)
check_data = first(check_loader)
fixed_image = check_data["fixed_hand"][0][0]
moving_image = check_data["moving_hand"][0][0]
print(f"moving_image shape: {moving_image.shape}")
print(f"fixed_image shape: {fixed_image.shape}")
plt.figure("check", (12, 6))
plt.subplot(1, 2, 1)
plt.title("moving_image")
plt.imshow(moving_image, cmap="gray")
plt.subplot(1, 2, 2)
plt.title("fixed_image")
plt.imshow(fixed_image, cmap="gray")
plt.show()
moving_image shape: torch.Size([64, 64]) fixed_image shape: torch.Size([64, 64])
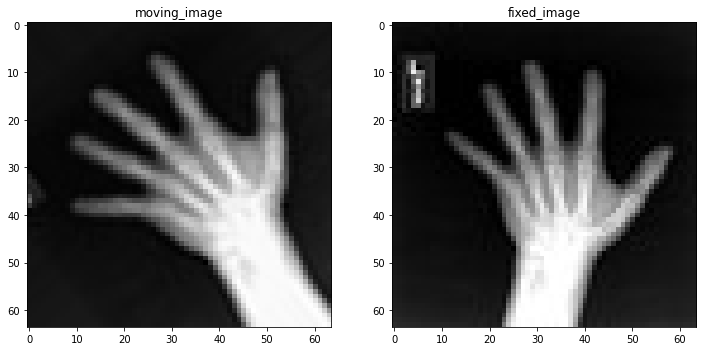
訓練パイプラインの作成
訓練ペアを capture して訓練プロセスを高速化するために CacheDataset を使用します。この訓練データは GlobalNet に供給されます、これは 画像レベルのアフィン変換パラメータを予測します。Warp 層は初期化されて訓練と推論の両方のために使用されます。
train_ds = CacheDataset(data=training_datadict[:1000], transform=train_transforms,
cache_rate=1.0, num_workers=4)
train_loader = DataLoader(train_ds, batch_size=16, shuffle=True, num_workers=2)
Loading dataset: 100%|██████████| 1000/1000 [00:01<00:00, 558.34it/s]
device = torch.device("cuda:0")
model = GlobalNet(
image_size=(64, 64),
spatial_dims=2,
in_channels=2, # moving and fixed
num_channel_initial=16,
depth=3).to(device)
image_loss = MSELoss()
if USE_COMPILED:
warp_layer = Warp(3, "border").to(device)
else:
warp_layer = Warp("bilinear", "border").to(device)
optimizer = torch.optim.Adam(model.parameters(), 1e-5)
訓練ループ
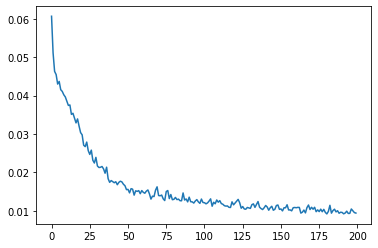
max_epochs = 200
epoch_loss_values = []
for epoch in range(max_epochs):
print("-" * 10)
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss, step = 0, 0
for batch_data in train_loader:
step += 1
optimizer.zero_grad()
moving = batch_data["moving_hand"].to(device)
fixed = batch_data["fixed_hand"].to(device)
ddf = model(torch.cat((moving, fixed), dim=1))
pred_image = warp_layer(moving, ddf)
loss = image_loss(pred_image, fixed)
loss.backward()
optimizer.step()
epoch_loss += loss.item()
# print(f"{step}/{len(train_ds) // train_loader.batch_size}, "
# f"train_loss: {loss.item():.4f}")
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
%matplotlib inline
plt.plot(epoch_loss_values)
幾つかの検証結果の視覚化
このセクションは moving vs fixed ハンドの未見の (i.e. 前に見てない) ペアのセットを作成して各ペアの間の変換を予測するためにネットワークを使用します。
val_ds = CacheDataset(data=training_datadict[2000:2500], transform=train_transforms,
cache_rate=1.0, num_workers=0)
val_loader = DataLoader(val_ds, batch_size=16, num_workers=0)
for batch_data in val_loader:
moving = batch_data["moving_hand"].to(device)
fixed = batch_data["fixed_hand"].to(device)
ddf = model(torch.cat((moving, fixed), dim=1))
pred_image = warp_layer(moving, ddf)
break
fixed_image = fixed.detach().cpu().numpy()[:, 0]
moving_image = moving.detach().cpu().numpy()[:, 0]
pred_image = pred_image.detach().cpu().numpy()[:, 0]
Loading dataset: 100%|██████████| 500/500 [00:00<00:00, 803.96it/s]
%matplotlib inline
batch_size = 5
plt.subplots(batch_size, 3, figsize=(8, 10))
for b in range(batch_size):
# moving image
plt.subplot(batch_size, 3, b * 3 + 1)
plt.axis('off')
plt.title("moving image")
plt.imshow(moving_image[b], cmap="gray")
# fixed image
plt.subplot(batch_size, 3, b * 3 + 2)
plt.axis('off')
plt.title("fixed image")
plt.imshow(fixed_image[b], cmap="gray")
# warped moving
plt.subplot(batch_size, 3, b * 3 + 3)
plt.axis('off')
plt.title("predicted image")
plt.imshow(pred_image[b], cmap="gray")
plt.axis('off')
plt.show()

以上
MONAI 1.0 : tutorials : モジュール – MedNIST データセットによる Autoencoder ネットワーク
MONAI 1.0 : tutorials : モジュール – MedNIST データセットによる Autoencoder ネットワーク (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 11/07/2022 (1.0.1)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP
MONAI 1.0 : tutorials : モジュール – MedNIST データセットによる Autoencoder ネットワーク
このチュートリアルは MONAI の autoencoder クラスを実演するために MedNIST ハンド CT スキャン・データセットを使用します。autoencoder は恒等エンコード/デコードで使用され (i.e. 貴方の入れたものが戻されるべきもの)、そしてぼかしとノイズの除去の使用方法として実演します。
このノートブックは画像のぼかし/ノイズ除去の目的で MONAI で autoencoeder の使用方法を示します。
学習目標
これは以下のステップを進みます :
- 遠隔ソースからデータをロードする
- 画像の辞書を作成するために lambda を使用する
- MONAI の組込み AutoEncoder を使用する
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[pillow, tqdm]"
1. インポートと設定
import logging
import os
import shutil
import sys
import tempfile
import random
import numpy as np
from tqdm import trange
import matplotlib.pyplot as plt
import torch
from skimage.util import random_noise
from monai.apps import download_and_extract
from monai.config import print_config
from monai.data import CacheDataset, DataLoader
from monai.networks.nets import AutoEncoder
from monai.transforms import (
EnsureChannelFirstD,
Compose,
LoadImageD,
RandFlipD,
RandRotateD,
RandZoomD,
ScaleIntensityD,
EnsureTypeD,
Lambda,
)
from monai.utils import set_determinism
print_config()
MONAI version: 0.6.0rc1+23.gc6793fd0
Numpy version: 1.20.3
Pytorch version: 1.9.0a0+c3d40fd
MONAI flags: HAS_EXT = True, USE_COMPILED = False
MONAI rev id: c6793fd0f316a448778d0047664aaf8c1895fe1c
Optional dependencies:
Pytorch Ignite version: 0.4.5
Nibabel version: 3.2.1
scikit-image version: 0.15.0
Pillow version: 8.2.0
Tensorboard version: 2.5.0
gdown version: 3.13.0
TorchVision version: 0.10.0a0
ITK version: 5.1.2
tqdm version: 4.53.0
lmdb version: 1.2.1
psutil version: 5.8.0
pandas version: 1.1.4
einops version: 0.3.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
logging.basicConfig(stream=sys.stdout, level=logging.INFO)
set_determinism(0)
device = torch.device("cuda" if torch.cuda.is_available() else "cpu")
# Create small visualisation function
def plot_ims(ims, shape=None, figsize=(10, 10), titles=None):
shape = (1, len(ims)) if shape is None else shape
plt.subplots(*shape, figsize=figsize)
for i, im in enumerate(ims):
plt.subplot(*shape, i + 1)
im = plt.imread(im) if isinstance(im, str) else torch.squeeze(im)
plt.imshow(im, cmap='gray')
if titles is not None:
plt.title(titles[i])
plt.axis('off')
plt.tight_layout()
plt.show()
データを取得する
MedMNIST データセットは TCIA, RSNA Bone Age チャレンジ と NIH Chest X-ray データセット からの様々なセットから集められました。
データセットは Dr. Bradley J. Erickson M.D., Ph.D. (Department of Radiology, Mayo Clinic) のお陰により Creative Commons CC BY-SA 4.0 ライセンス のもとで利用可能になっています。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical
resource = "https://github.com/Project-MONAI/MONAI-extra-test-data/releases/download/0.8.1/MedNIST.tar.gz"
md5 = "0bc7306e7427e00ad1c5526a6677552d"
compressed_file = os.path.join(root_dir, "MedNIST.tar.gz")
data_dir = os.path.join(root_dir, "MedNIST")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
# scan_type could be AbdomenCT BreastMRI CXR ChestCT Hand HeadCT
scan_type = "Hand"
im_dir = os.path.join(data_dir, scan_type)
all_filenames = [os.path.join(im_dir, filename)
for filename in os.listdir(im_dir)]
random.shuffle(all_filenames)
# Visualise a few of them
rand_images = np.random.choice(all_filenames, 8, replace=False)
plot_ims(rand_images, shape=(2, 4))

# Split into training and testing
test_frac = 0.2
num_test = int(len(all_filenames) * test_frac)
num_train = len(all_filenames) - num_test
train_datadict = [{"im": fname} for fname in all_filenames[:num_train]]
test_datadict = [{"im": fname} for fname in all_filenames[-num_test:]]
print(f"total number of images: {len(all_filenames)}")
print(f"number of images for training: {len(train_datadict)}")
print(f"number of images for testing: {len(test_datadict)}")
total number of images: 10000 number of images for training: 8000 number of images for testing: 2000
3. 画像変換チェインを作成する
画像のぼやけ/ノイズを除去する autoencoder を訓練するため、劣化画像をエンコーダに渡すことを望みますが、損失関数では、元の劣化していないバージョンとの比較を行ないます。この意味で、エンコードとデコードステップが劣化を除去できたときに、損失関数は最小化されます。
画像の一つのバージョンが劣化していて他方がそうではないという事実以外に、それらが同一であるようにすることを望みます、これは同じ変換から生成される必要があることを意味します。これを行なう最も簡単な方法は辞書変換を使うことです、そこでは最後に、3 つの画像 – オリジナル、ガウスぼかしとノイズのある (画像) を含む辞書を返す lambda 関数を持ちます。
NoiseLambda = Lambda(lambda d: {
"orig": d["im"],
"gaus": torch.tensor(
random_noise(d["im"], mode='gaussian'), dtype=torch.float32),
"s&p": torch.tensor(random_noise(d["im"], mode='s&p', salt_vs_pepper=0.1)),
})
train_transforms = Compose(
[
LoadImageD(keys=["im"]),
EnsureChannelFirstD(keys=["im"]),
ScaleIntensityD(keys=["im"]),
RandRotateD(keys=["im"], range_x=np.pi / 12, prob=0.5, keep_size=True),
RandFlipD(keys=["im"], spatial_axis=0, prob=0.5),
RandZoomD(keys=["im"], min_zoom=0.9, max_zoom=1.1, prob=0.5),
EnsureTypeD(keys=["im"]),
NoiseLambda,
]
)
test_transforms = Compose(
[
LoadImageD(keys=["im"]),
EnsureChannelFirstD(keys=["im"]),
ScaleIntensityD(keys=["im"]),
EnsureTypeD(keys=["im"]),
NoiseLambda,
]
)
データセットとデータローダの作成
データを保持して訓練の間にバッチを提示する
batch_size = 300
num_workers = 10
train_ds = CacheDataset(train_datadict, train_transforms,
num_workers=num_workers)
train_loader = DataLoader(train_ds, batch_size=batch_size,
shuffle=True, num_workers=num_workers)
test_ds = CacheDataset(test_datadict, test_transforms, num_workers=num_workers)
test_loader = DataLoader(test_ds, batch_size=batch_size,
shuffle=True, num_workers=num_workers)
100%|██████████| 8000/8000 [00:02<00:00, 2698.01it/s] 100%|██████████| 2000/2000 [00:02<00:00, 904.36it/s]
# Get image original and its degraded versions
def get_single_im(ds):
loader = torch.utils.data.DataLoader(
ds, batch_size=1, num_workers=10, shuffle=True)
itera = iter(loader)
return next(itera)
data = get_single_im(train_ds)
plot_ims([data['orig'], data['gaus'], data['s&p']],
titles=['orig', 'Gaussian', 's&p'])
def train(dict_key_for_training, max_epochs=10, learning_rate=1e-3):
model = AutoEncoder(
spatial_dims=2,
in_channels=1,
out_channels=1,
channels=(4, 8, 16, 32),
strides=(2, 2, 2, 2),
).to(device)
# Create loss fn and optimiser
loss_function = torch.nn.MSELoss()
optimizer = torch.optim.Adam(model.parameters(), learning_rate)
epoch_loss_values = []
t = trange(
max_epochs,
desc=f"{dict_key_for_training} -- epoch 0, avg loss: inf", leave=True)
for epoch in t:
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step += 1
inputs = batch_data[dict_key_for_training].to(device)
optimizer.zero_grad()
outputs = model(inputs)
loss = loss_function(outputs, batch_data['orig'].to(device))
loss.backward()
optimizer.step()
epoch_loss += loss.item()
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
t.set_description(
f"{dict_key_for_training} -- epoch {epoch + 1}"
+ f", average loss: {epoch_loss:.4f}")
return model, epoch_loss_values
max_epochs = 50
training_types = ['orig', 'gaus', 's&p']
models = []
epoch_losses = []
for training_type in training_types:
model, epoch_loss = train(training_type, max_epochs=max_epochs)
models.append(model)
epoch_losses.append(epoch_loss)
orig -- epoch 30, average loss: 0.0110: 60%|██████ | 30/50 [01:41<01:08, 3.40s/it]
plt.figure()
plt.title("Epoch Average Loss")
plt.xlabel("epoch")
for y, label in zip(epoch_losses, training_types):
x = list(range(1, len(y) + 1))
line, = plt.plot(x, y)
line.set_label(label)
plt.legend()
data = get_single_im(test_ds)
recons = []
for model, training_type in zip(models, training_types):
im = data[training_type]
recon = model(im.to(device)).detach().cpu()
recons.append(recon)
plot_ims(
[data['orig'], data['gaus'], data['s&p']] + recons,
titles=['orig', 'Gaussian', 'S&P'] +
["recon w/\n" + x for x in training_types],
shape=(2, len(training_types)))
データディレクトリのクリーンアップ
一時ディレクトリが使用された場合ディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
MONAI 1.0 : チュートリアル
MONAI 1.0 : チュートリアル
翻訳 : (株)クラスキャット セールスインフォメーション
更新日時 : 01/19/2023 (1.1.0)
作成日時 : 11/02/2022 (1.0.1)

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
- 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション
- sales-info@classcat.com ; Web: www.classcat.com ; ClassCatJP

MONAI 1.0 : チュートリアル
2D 分類
- MedNIST データセットによる医用画像分類 (2d_classification/mednist_tutorial) (11/02/2022)
3D セグメンテーション
- 脳腫瘍 3D セグメンテーション (brats_segmentation_3d) (01/18/2023)
- 脾臓 3D セグメンテーション (spleen_segmentation_3d) (01/19/2023)
2D レジストレーション
- 2D XRay レジストレーション・デモ (2d_registration/registration_mednist) (11/08/2022)
モジュール
- MedNIST データセットによる Autoencoder ネットワーク (modules/autoencoder_mednist) (11/07/2022)
- MedNIST で GAN (modules/mednist_GAN_tutorial) (11/08/2022)
- GAN ワークフロー・エンジン (辞書版) (mednist_GAN_workflow_dict) (11/13/2022)
- 2D 画像変換デモ (transforms_demo_2d) (11/11/2021)
以上
