MONAI 0.7 : tutorials : 配備 – BentoML による MedNIST 分類器の配備 (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 10/19/2021 (0.7.0)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。
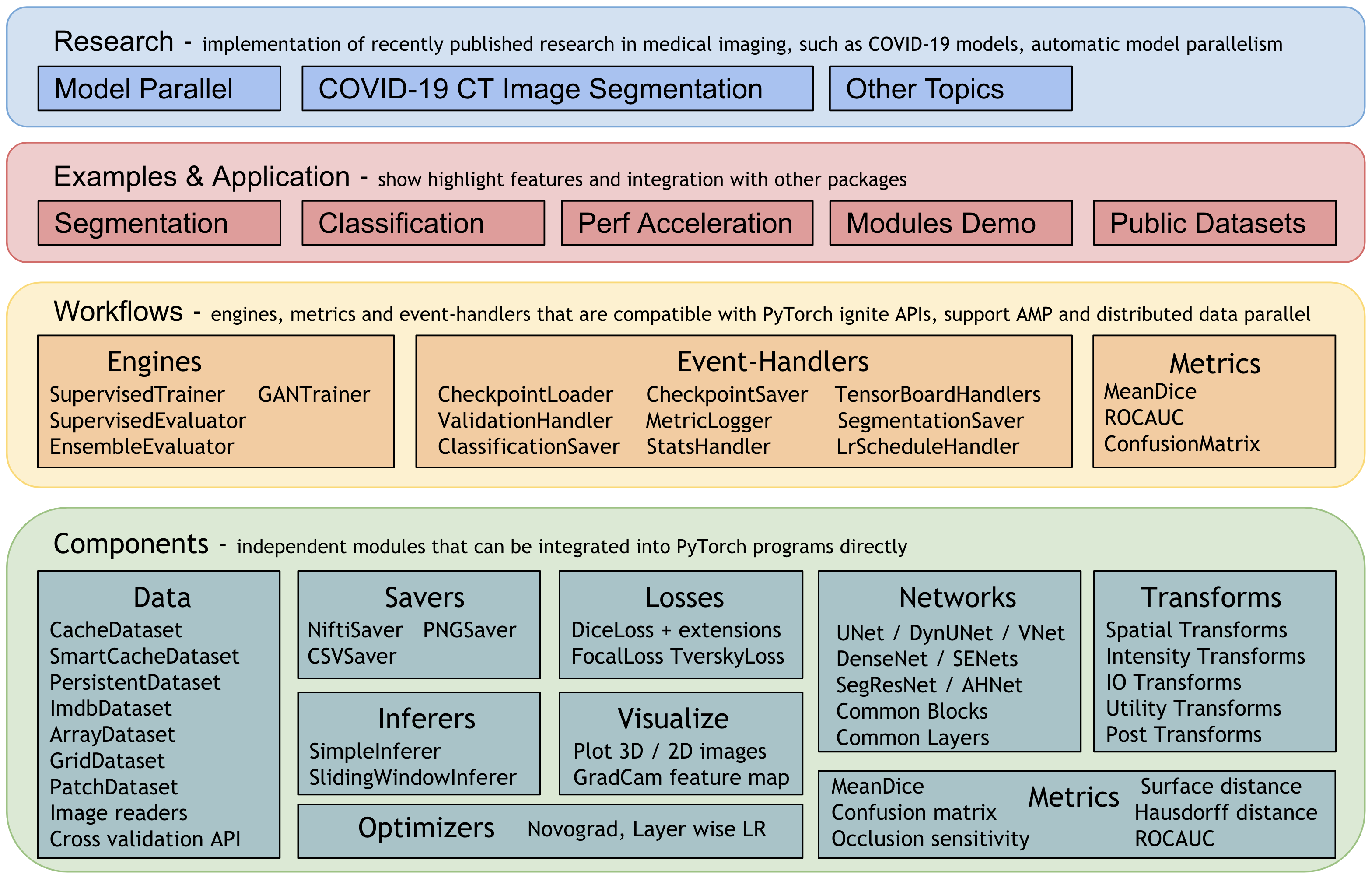
MONAI 0.7 : tutorials : 配備 – BentoML による MedNIST 分類器の配備
これは MONAI ネットワークを訓練して BentoML を web サーバとして使用して配備するサンプルです、BentoML レポジトリをローカルで使用するかコンテナサービスとして使用します。
このノートブックは BentoML を使用して訓練済みモデルをアーティファクトにパッケージ化するプロセスを実演します、これは推論を実行するローカルプログラムとして、同じことを行なう web サービスとして、そして Docker コンテナ化された web サービスとして実行できます。BentoML は AWS や Azure のような既存のプラットフォームでモデルを配備する様々な方法を提供しますが、ここではローカル配備にフォーカスします、研究者はこれを行なう傾向にあるためです。このチュートリアルは ここの MONAI チュートリアル のような MedNIST 分類器を訓練してから BentoML チュートリアル で説明されているパッケージ化を行ないます。
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[pillow, tqdm]"
!python -c "import bentoml" || pip install -q bentoml
インポートのセットアップ
# Copyright 2020 MONAI Consortium
# Licensed under the Apache License, Version 2.0 (the "License");
# you may not use this file except in compliance with the License.
# You may obtain a copy of the License at
# http://www.apache.org/licenses/LICENSE-2.0
# Unless required by applicable law or agreed to in writing, software
# distributed under the License is distributed on an "AS IS" BASIS,
# WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied.
# See the License for the specific language governing permissions and
# limitations under the License.
import os
import shutil
import tempfile
import glob
import PIL.Image
import torch
import numpy as np
from ignite.engine import Events
from monai.apps import download_and_extract
from monai.config import print_config
from monai.networks.nets import DenseNet121
from monai.engines import SupervisedTrainer
from monai.transforms import (
AddChannel,
Compose,
LoadImage,
RandFlip,
RandRotate,
RandZoom,
ScaleIntensity,
EnsureType,
)
from monai.utils import set_determinism
set_determinism(seed=0)
print_config()
MONAI version: 0.4.0+119.g9898a89
Numpy version: 1.19.2
Pytorch version: 1.7.1
MONAI flags: HAS_EXT = False, USE_COMPILED = False
MONAI rev id: 9898a89d24364a9be3525d066a7492adf00b9e6b
Optional dependencies:
Pytorch Ignite version: 0.4.2
Nibabel version: 3.2.1
scikit-image version: 0.18.1
Pillow version: 8.1.0
Tensorboard version: 2.4.1
gdown version: 3.12.2
TorchVision version: 0.8.2
ITK version: 5.1.2
tqdm version: 4.56.0
lmdb version: 1.0.0
psutil version: 5.8.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データセットをダウンロードする
MedMNIST データセットは TCIA, RSNA Bone Age チャレンジ と NIH Chest X-ray データセット からの様々なセットから集められました。
データセットは Dr. Bradley J. Erickson M.D., Ph.D. (Department of Radiology, Mayo Clinic) のお陰により Creative Commons CC BY-SA 4.0 ライセンス のもとで利用可能になっています。
MedNIST データセットを使用する場合、出典を明示してください。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
resource = "https://drive.google.com/uc?id=1QsnnkvZyJPcbRoV_ArW8SnE1OTuoVbKE"
md5 = "0bc7306e7427e00ad1c5526a6677552d"
compressed_file = os.path.join(root_dir, "MedNIST.tar.gz")
data_dir = os.path.join(root_dir, "MedNIST")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
MedNIST.tar.gz: 0.00B [00:00, ?B/s] /tmp/tmpxxp5z205 MedNIST.tar.gz: 59.0MB [00:04, 15.4MB/s] downloaded file: /tmp/tmpxxp5z205/MedNIST.tar.gz. Verified 'MedNIST.tar.gz', md5: 0bc7306e7427e00ad1c5526a6677552d. Verified 'MedNIST.tar.gz', md5: 0bc7306e7427e00ad1c5526a6677552d.
subdirs = sorted(glob.glob(f"{data_dir}/*/"))
class_names = [os.path.basename(sd[:-1]) for sd in subdirs]
image_files = [glob.glob(f"{sb}/*") for sb in subdirs]
image_files_list = sum(image_files, [])
image_class = sum(([i] * len(f) for i, f in enumerate(image_files)), [])
image_width, image_height = PIL.Image.open(image_files_list[0]).size
print(f"Label names: {class_names}")
print(f"Label counts: {list(map(len, image_files))}")
print(f"Total image count: {len(image_class)}")
print(f"Image dimensions: {image_width} x {image_height}")
Label names: ['AbdomenCT', 'BreastMRI', 'CXR', 'ChestCT', 'Hand', 'HeadCT'] Label counts: [10000, 8954, 10000, 10000, 10000, 10000] Total image count: 58954 Image dimensions: 64 x 64
セットアップと訓練
ここでは変換シークエンスを作成してネットワークを訓練します、検証とテストはこれが実際に動作することを私達は知っていてそしてここでは必要ないので省略します :
train_transforms = Compose(
[
LoadImage(image_only=True),
AddChannel(),
ScaleIntensity(),
RandRotate(range_x=np.pi / 12, prob=0.5, keep_size=True),
RandFlip(spatial_axis=0, prob=0.5),
RandZoom(min_zoom=0.9, max_zoom=1.1, prob=0.5),
EnsureType(),
]
)
class MedNISTDataset(torch.utils.data.Dataset):
def __init__(self, image_files, labels, transforms):
self.image_files = image_files
self.labels = labels
self.transforms = transforms
def __len__(self):
return len(self.image_files)
def __getitem__(self, index):
return self.transforms(self.image_files[index]), self.labels[index]
# just one dataset and loader, we won't bother with validation or testing
train_ds = MedNISTDataset(image_files_list, image_class, train_transforms)
train_loader = torch.utils.data.DataLoader(train_ds, batch_size=300, shuffle=True, num_workers=10)
device = torch.device("cuda:0")
net = DenseNet121(spatial_dims=2, in_channels=1, out_channels=len(class_names)).to(device)
loss_function = torch.nn.CrossEntropyLoss()
opt = torch.optim.Adam(net.parameters(), 1e-5)
max_epochs = 5
def _prepare_batch(batch, device, non_blocking):
return tuple(b.to(device) for b in batch)
trainer = SupervisedTrainer(device, max_epochs, train_loader, net, opt, loss_function, prepare_batch=_prepare_batch)
@trainer.on(Events.EPOCH_COMPLETED)
def _print_loss(engine):
print(f"Epoch {engine.state.epoch}/{engine.state.max_epochs} Loss: {engine.state.output[0]['loss']}")
trainer.run()
Epoch 1/5 Loss: 0.231450617313385 Epoch 2/5 Loss: 0.07256477326154709 Epoch 3/5 Loss: 0.04309789836406708 Epoch 4/5 Loss: 0.04549304023385048 Epoch 5/5 Loss: 0.025731785222887993
ここでネットワークが Torchscript オブジェクトとしてセーブされますが後で見るようこれは必要ありません。
torch.jit.script(net).save("classifier.zip")
BentoML セットアップ
BentoML はサービスリクエストをメソッド呼び出しとしてラップする API を通してプラットフォームを提供します。これは明らかに Flask が動作する方法と似ていますが (これはここで使用される基礎技術の一つです)、これの上にはネットワーク (アーティファクト) のストア、リクエストの IO コンポーネントの処理、そしてデータのキャッシュのための様々な機能が提供されます。私達が提供する必要があるものは望むサービスを表わすスクリプトファイルで、BentoML は提供するアーティファクトと一緒にこれを取得して別の場所にストアします、これはローカルで実行したりサーバにアップロードすることができます (Docker レジストリのようなものです)。
下のスクリプトは MONAI コードを含む API を作成します。変換シークエンスはデータストリームを画像に変えるために特殊な読み取り変換 (= read Transform) を必要としますが、それ以外は訓練のために上で使用されたようなコードです。ネットワークはアーティファクトとしてストアされ、これは実際には BentoML バンドルでストアされた重みです。これは実行時に自動的にロードされますが、望むならば代わりに Torchscript モデルをロードすることもできるでしょう、特に MONAI コードに依存しない API を望む場合には。
スクリプトは最初にファイルに書き出される必要があります :
%%writefile mednist_classifier_bentoml.py
from typing import BinaryIO, List
import numpy as np
from PIL import Image
import torch
from monai.transforms import (
AddChannel,
Compose,
Transform,
ScaleIntensity,
EnsureType,
)
import bentoml
from bentoml.frameworks.pytorch import PytorchModelArtifact
from bentoml.adapters import FileInput, JsonOutput
from bentoml.utils import cached_property
MEDNIST_CLASSES = ["AbdomenCT", "BreastMRI", "CXR", "ChestCT", "Hand", "HeadCT"]
class LoadStreamPIL(Transform):
"""Load an image file from a data stream using PIL."""
def __init__(self, mode=None):
self.mode = mode
def __call__(self, stream):
img = Image.open(stream)
if self.mode is not None:
img = img.convert(mode=self.mode)
return np.array(img)
@bentoml.env(pip_packages=["torch", "numpy", "monai", "pillow"])
@bentoml.artifacts([PytorchModelArtifact("classifier")])
class MedNISTClassifier(bentoml.BentoService):
@cached_property
def transform(self):
return Compose([LoadStreamPIL("L"), AddChannel(), ScaleIntensity(), EnsureType()])
@bentoml.api(input=FileInput(), output=JsonOutput(), batch=True)
def predict(self, file_streams: List[BinaryIO]) -> List[str]:
img_tensors = list(map(self.transform, file_streams))
batch = torch.stack(img_tensors).float()
with torch.no_grad():
outputs = self.artifacts.classifier(batch)
_, output_classes = outputs.max(dim=1)
return [MEDNIST_CLASSES[oc] for oc in output_classes]
Overwriting mednist_classifier_bentoml.py
今はスクリプトがロードされて分類器アーティファクトはネットワーク状態とともにパックされます。そしてこれはローカルマシンのレポジトリ・ディレクトリに保存されます :
from mednist_classifier_bentoml import MedNISTClassifier # noqa: E402
bento_svc = MedNISTClassifier()
bento_svc.pack('classifier', net.cpu().eval())
saved_path = bento_svc.save()
print(saved_path)
[2021-03-02 00:39:04,202] WARNING - BentoML by default does not include spacy and torchvision package when using PytorchModelArtifact. To make sure BentoML bundle those packages if they are required for your model, either import those packages in BentoService definition file or manually add them via `@env(pip_packages=['torchvision'])` when defining a BentoService [2021-03-02 00:39:04,204] WARNING - pip package requirement torch already exist [2021-03-02 00:39:05,494] INFO - BentoService bundle 'MedNISTClassifier:20210302003904_AC4A5D' saved to: /home/localek10/bentoml/repository/MedNISTClassifier/20210302003904_AC4A5D /home/localek10/bentoml/repository/MedNISTClassifier/20210302003904_AC4A5D
このレポジトリの内容を見ることができます、これはコードとセットアップ・スクリプトを含みます :
!ls -l {saved_path}
total 44 -rwxr--r-- 1 localek10 bioeng 2411 Mar 2 00:39 bentoml-init.sh -rw-r--r-- 1 localek10 bioeng 875 Mar 2 00:39 bentoml.yml -rwxr--r-- 1 localek10 bioeng 699 Mar 2 00:39 docker-entrypoint.sh -rw-r--r-- 1 localek10 bioeng 1205 Mar 2 00:39 Dockerfile -rw-r--r-- 1 localek10 bioeng 70 Mar 2 00:39 environment.yml -rw-r--r-- 1 localek10 bioeng 72 Mar 2 00:39 MANIFEST.in drwxr-xr-x 4 localek10 bioeng 4096 Mar 2 00:39 MedNISTClassifier -rw-r--r-- 1 localek10 bioeng 5 Mar 2 00:39 python_version -rw-r--r-- 1 localek10 bioeng 298 Mar 2 00:39 README.md -rw-r--r-- 1 localek10 bioeng 69 Mar 2 00:39 requirements.txt -rw-r--r-- 1 localek10 bioeng 1691 Mar 2 00:39 setup.py
このレポジトリはストアされたプログラムのように実行できます、そこでは使用したい名前と API 名 (“predict”) によりそれを起動してファイルとして入力を提供します :
!bentoml run MedNISTClassifier:latest predict --input-file {image_files[0][0]}
[2021-03-02 00:39:16,999] INFO - Getting latest version MedNISTClassifier:20210302003904_AC4A5D
[2021-03-02 00:39:19,508] WARNING - BentoML by default does not include spacy and torchvision package when using PytorchModelArtifact. To make sure BentoML bundle those packages if they are required for your model, either import those packages in BentoService definition file or manually add them via `@env(pip_packages=['torchvision'])` when defining a BentoService
[2021-03-02 00:39:19,508] WARNING - pip package requirement torch already exist
[2021-03-02 00:39:20,329] INFO - {'service_name': 'MedNISTClassifier', 'service_version': '20210302003904_AC4A5D', 'api': 'predict', 'task': {'data': {'uri': 'file:///tmp/tmphl16qkwk/MedNIST/AbdomenCT/006160.jpeg', 'name': '006160.jpeg'}, 'task_id': '6d4680de-f719-4e04-abde-00c7d8a6110d', 'cli_args': ('--input-file', '/tmp/tmphl16qkwk/MedNIST/AbdomenCT/006160.jpeg'), 'inference_job_args': {}}, 'result': {'data': '"Hand"', 'http_status': 200, 'http_headers': (('Content-Type', 'application/json'),)}, 'request_id': '6d4680de-f719-4e04-abde-00c7d8a6110d'}
"Hand"
サービスはまた Flask web サーバでも実行できます。以下のスクリプトはサービスを開始し、進むのを待ち、予測を得るために POST リクエストとしてテストファイルを送るために curl を使用して、そしてサーバを kill します :
%%bash -s {image_files[0][0]}
# filename passed in as an argument to the cell
test_file=$1
# start the Flask-based server, sending output to /dev/null for neatness
bentoml serve --port=8000 MedNISTClassifier:latest &> /dev/null &
# recall the PID of the server and wait for it to start
lastpid=$!
sleep 5
# send the test file using curl and capture the returned string
result=$(curl -s -X POST "http://127.0.0.1:8000/predict" -F image=@$test_file)
# kill the server
kill $lastpid
echo "Prediction: $result"
Prediction: "AbdomenCT"
The service can be packaged as a Docker container to be started elsewhere as a server:
!bentoml containerize MedNISTClassifier:latest -t mednist-classifier:latest
[2021-03-02 00:40:48,846] INFO - Getting latest version MedNISTClassifier:20210302003904_AC4A5D Found Bento: /home/localek10/bentoml/repository/MedNISTClassifier/20210302003904_AC4A5D Containerizing MedNISTClassifier:20210302003904_AC4A5D with local YataiService and docker daemon from local environment\WARNING: No swap limit support |Build container image: mednist-classifier:latest
!docker image ls
REPOSITORY TAG IMAGE ID CREATED SIZE mednist-classifier latest 326ab3f07478 15 seconds ago 2.94GB <none> <none> 87e9c5c97297 2 days ago 2.94GB <none> <none> cb62f45a9163 2 days ago 1.14GB bentoml/model-server 0.11.0-py38 387830631375 6 weeks ago 1.14GB sshtest latest 1be604ad1135 3 months ago 225MB ubuntu 20.04 9140108b62dc 5 months ago 72.9MB ubuntu latest 9140108b62dc 5 months ago 72.9MB nvcr.io/nvidia/pytorch 20.09-py3 86042df4bd3c 5 months ago 11.1GB pytorch/pytorch 1.6.0-cuda10.1-cudnn7-runtime 6a2d656bcf94 7 months ago 3.47GB pytorch/pytorch latest 6a2d656bcf94 7 months ago 3.47GB python 3.7 22c70bba8283 7 months ago 920MB ubuntu 16.04 c522ac0d6194 7 months ago 126MB python 3.7-alpine 6a5ca85ed89b 9 months ago 72.5MB alpine 3.12 a24bb4013296 9 months ago 5.57MB hello-world latest bf756fb1ae65 14 months ago 13.3kB
if directory is None:
shutil.rmtree(root_dir)
以上

