MONAI 0.7 : tutorials : 3D セグメンテーション – 脳腫瘍 3D セグメンテーション (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 11/08/2021 (0.7.0)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- テレワーク & オンライン授業を支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
- ウェビナー運用には弊社製品「ClassCat® Webinar」を利用しています。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
| 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション |
| E-Mail:sales-info@classcat.com ; WebSite: https://www.classcat.com/ ; Facebook |
MONAI 0.7 : tutorials : 3D セグメンテーション – 脳腫瘍 3D セグメンテーション
このチュートリアルは MSD 脳腫瘍データセットに基づくマルチラベル・タスクの訓練ワークフローを構築する方法を示します。
このチュートリアルはマルチラベル・セグメンテーション・タスクの訓練ワークフローを構築する方法を示します。
そしてそれは以下の機能を含みます :
- 辞書形式データのための変換。
- MONAI 変換 API に従って新しい変換を定義する。
- メタデータと共に Nifti 画像をロードし、画像のリストをロードしてそれらをスタックする。
- データ増強のために強度をランダムに調整する。
- 訓練と検証を高速化する Cache IO と変換。
- 3D セグメンテーション・タスクのための 3D SegResNet モデル, Dice 損失関数, 平均 Dice メトリック。
- 再現性のための決定論的訓練。
データセットは http://medicaldecathlon.com/ に由来します。
ターゲット: Gliomas segmentation necrotic/active tumour and oedema
Modality: Multimodal multisite MRI データ (FLAIR, T1w, T1gd,T2w)
サイズ: 750 4D volumes (484 訓練 + 266 テスト)
ソース: BRATS 2016 と 2017 データセット。
チャレンジ: Complex and heterogeneously-located targets
下図は、様々なモダリティでアノテートされている腫瘍部分領域の画像パッチ (左上) とデータセット全体のための最終的なラベル (右) を示します。(図は BraTS IEEE TMI 論文 から引用)
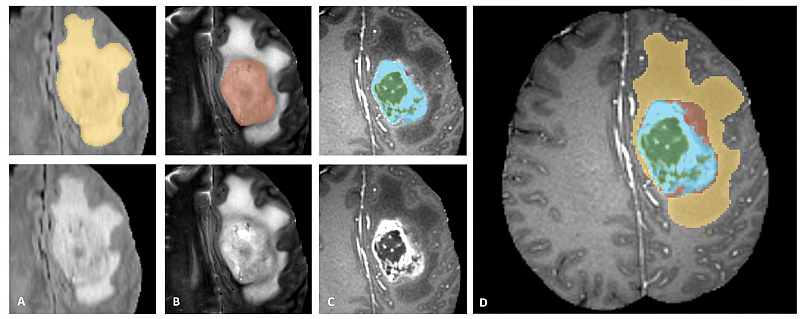
画像パッチは左から右へ以下を示します :
- T2-FLAIR で見える腫瘍全体 (黄色) (Fig.A)。
- T2 で見える腫瘍のコア (赤色) (Fig. B)。
- T1Gd で見える enhancing 腫瘍構造 (ライトブルー)、これはコアの嚢胞 (のうほう) (= cystic) / 壊死 (=necrotic) 成分 (緑色) を取り囲んでいます (Fig. C)。
- セグメンテーションは 腫瘍部分領域の最終的なラベル (Fig.D) を生成するために組み合わされます : 浮腫 (= edema) (黄色), non-enhancing ソリッドコア (赤色), 嚢胞/壊死コア (緑色), enhancing コア (青色) です。
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[nibabel, tqdm]"
!python -c "import matplotlib" || pip install -q matplotlib
#!pip install pytorch-ignite
%matplotlib inline
インポートのセットアップ
# Copyright 2020 MONAI Consortium
# Licensed under the Apache License, Version 2.0 (the "License");
# you may not use this file except in compliance with the License.
# You may obtain a copy of the License at
# http://www.apache.org/licenses/LICENSE-2.0
# Unless required by applicable law or agreed to in writing, software
# distributed under the License is distributed on an "AS IS" BASIS,
# WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied.
# See the License for the specific language governing permissions and
# limitations under the License.
import os
import shutil
import tempfile
import time
import matplotlib.pyplot as plt
import numpy as np
from monai.apps import DecathlonDataset
from monai.config import print_config
from monai.data import DataLoader, decollate_batch
from monai.handlers.utils import from_engine
from monai.losses import DiceLoss
from monai.inferers import sliding_window_inference
from monai.metrics import DiceMetric
from monai.networks.nets import SegResNet
from monai.transforms import (
Activations,
Activationsd,
AsDiscrete,
AsDiscreted,
Compose,
Invertd,
LoadImaged,
MapTransform,
NormalizeIntensityd,
Orientationd,
RandFlipd,
RandScaleIntensityd,
RandShiftIntensityd,
RandSpatialCropd,
Spacingd,
EnsureChannelFirstd,
EnsureTyped,
EnsureType,
)
from monai.utils import set_determinism
import torch
print_config()
MONAI version: 0.4.0+618.g69b44596
Numpy version: 1.20.3
Pytorch version: 1.9.0a0+c3d40fd
MONAI flags: HAS_EXT = False, USE_COMPILED = False
MONAI rev id: 69b4459650fb6943b9e729e724254d2db2b2a1f2
Optional dependencies:
Pytorch Ignite version: 0.4.5
Nibabel version: 3.2.1
scikit-image version: 0.15.0
Pillow version: 8.3.1
Tensorboard version: 2.5.0
gdown version: 3.13.0
TorchVision version: 0.10.0a0
tqdm version: 4.53.0
lmdb version: 1.2.1
psutil version: 5.8.0
pandas version: 1.1.4
einops version: 0.3.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。これは結果をセーブしてダウンロードを再利用することを可能にします。指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical
再現性のために決定論的訓練を設定する
set_determinism(seed=0)
脳腫瘍のラベルを変換するための新しい変換を定義する
ここでは多クラスラベルを One-Hot 形式のマルチラベルのセグメンテーション・タスクに変換します。
class ConvertToMultiChannelBasedOnBratsClassesd(MapTransform):
"""
Convert labels to multi channels based on brats classes:
label 1 is the peritumoral edema
label 2 is the GD-enhancing tumor
label 3 is the necrotic and non-enhancing tumor core
The possible classes are TC (Tumor core), WT (Whole tumor)
and ET (Enhancing tumor).
"""
def __call__(self, data):
d = dict(data)
for key in self.keys:
result = []
# merge label 2 and label 3 to construct TC
result.append(np.logical_or(d[key] == 2, d[key] == 3))
# merge labels 1, 2 and 3 to construct WT
result.append(
np.logical_or(
np.logical_or(d[key] == 2, d[key] == 3), d[key] == 1
)
)
# label 2 is ET
result.append(d[key] == 2)
d[key] = np.stack(result, axis=0).astype(np.float32)
return d
訓練と検証のための変換のセットアップ
train_transform = Compose(
[
# load 4 Nifti images and stack them together
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
Orientationd(keys=["image", "label"], axcodes="RAS"),
RandSpatialCropd(keys=["image", "label"], roi_size=[224, 224, 144], random_size=False),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=0),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=1),
RandFlipd(keys=["image", "label"], prob=0.5, spatial_axis=2),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
RandScaleIntensityd(keys="image", factors=0.1, prob=1.0),
RandShiftIntensityd(keys="image", offsets=0.1, prob=1.0),
EnsureTyped(keys=["image", "label"]),
]
)
val_transform = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys="image"),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(
keys=["image", "label"],
pixdim=(1.0, 1.0, 1.0),
mode=("bilinear", "nearest"),
),
Orientationd(keys=["image", "label"], axcodes="RAS"),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
EnsureTyped(keys=["image", "label"]),
]
)
DecathlonDataset でデータを素早くロードする
ここではデータセットを自動的にダウンロードして抽出するために DecathlonDataset を使用します。それは MONAI CacheDataset を継承し、より少ないメモリを使用したい場合には、訓練のために N 項目をキャッシュするために cache_num=N を設定して検証のために総ての項目をキャッシュするために default args を使用できます、それはメモリサイズに依存します。
# here we don't cache any data in case out of memory issue
train_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=train_transform,
section="training",
download=True,
cache_rate=0.0,
num_workers=4,
)
train_loader = DataLoader(train_ds, batch_size=1, shuffle=True, num_workers=4)
val_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=val_transform,
section="validation",
download=False,
cache_rate=0.0,
num_workers=4,
)
val_loader = DataLoader(val_ds, batch_size=1, shuffle=False, num_workers=4)
Verified 'Task01_BrainTumour.tar', md5: 240a19d752f0d9e9101544901065d872. File exists: /workspace/data/medical/Task01_BrainTumour.tar, skipped downloading. Non-empty folder exists in /workspace/data/medical/Task01_BrainTumour, skipped extracting.
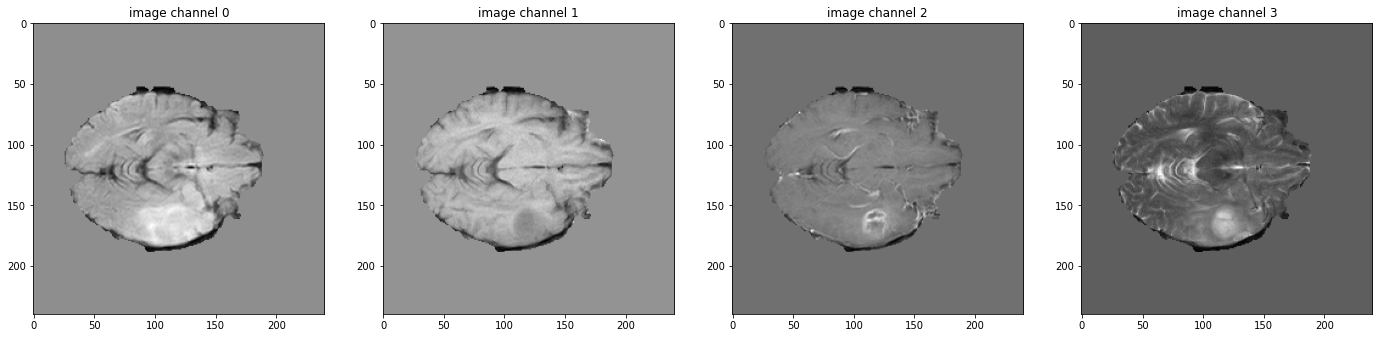
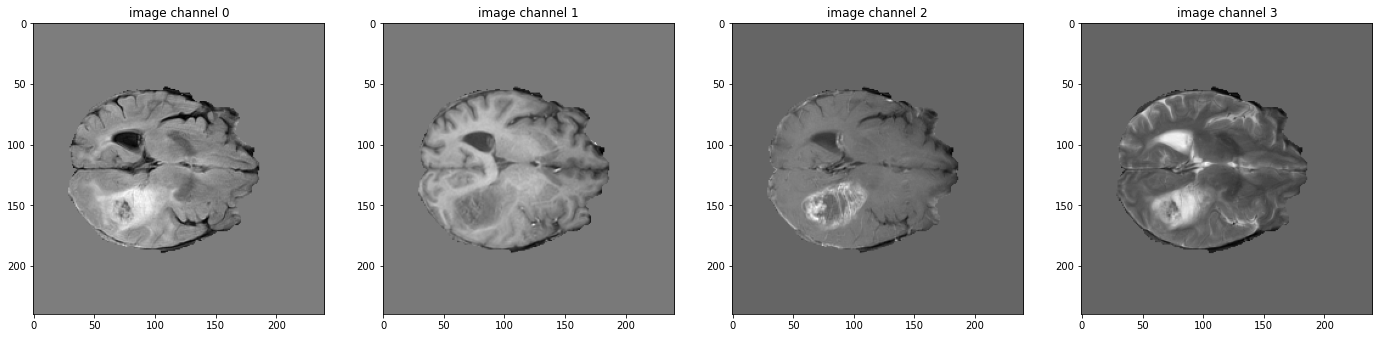
データ shape を確認して可視化する
# pick one image from DecathlonDataset to visualize and check the 4 channels
print(f"image shape: {val_ds[2]['image'].shape}")
plt.figure("image", (24, 6))
for i in range(4):
plt.subplot(1, 4, i + 1)
plt.title(f"image channel {i}")
plt.imshow(val_ds[2]["image"][i, :, :, 60].detach().cpu(), cmap="gray")
plt.show()
# also visualize the 3 channels label corresponding to this image
print(f"label shape: {val_ds[2]['label'].shape}")
plt.figure("label", (18, 6))
for i in range(3):
plt.subplot(1, 3, i + 1)
plt.title(f"label channel {i}")
plt.imshow(val_ds[2]["label"][i, :, :, 60].detach().cpu())
plt.show()
image shape: torch.Size([4, 240, 240, 155])

モデル, 損失, Optimizer を作成する
max_epochs = 300
val_interval = 1
VAL_AMP = True
# standard PyTorch program style: create SegResNet, DiceLoss and Adam optimizer
device = torch.device("cuda:0")
model = SegResNet(
blocks_down=[1, 2, 2, 4],
blocks_up=[1, 1, 1],
init_filters=16,
in_channels=4,
out_channels=3,
dropout_prob=0.2,
).to(device)
loss_function = DiceLoss(smooth_nr=0, smooth_dr=1e-5, squared_pred=True, to_onehot_y=False, sigmoid=True)
optimizer = torch.optim.Adam(model.parameters(), 1e-4, weight_decay=1e-5)
lr_scheduler = torch.optim.lr_scheduler.CosineAnnealingLR(optimizer, T_max=max_epochs)
dice_metric = DiceMetric(include_background=True, reduction="mean")
dice_metric_batch = DiceMetric(include_background=True, reduction="mean_batch")
post_trans = Compose(
[EnsureType(), Activations(sigmoid=True), AsDiscrete(threshold_values=True)]
)
# define inference method
def inference(input):
def _compute(input):
return sliding_window_inference(
inputs=input,
roi_size=(240, 240, 160),
sw_batch_size=1,
predictor=model,
overlap=0.5,
)
if VAL_AMP:
with torch.cuda.amp.autocast():
return _compute(input)
else:
return _compute(input)
# use amp to accelerate training
scaler = torch.cuda.amp.GradScaler()
# enable cuDNN benchmark
torch.backends.cudnn.benchmark = True
典型的な PyTorch 訓練プロセスの実行
best_metric = -1
best_metric_epoch = -1
best_metrics_epochs_and_time = [[], [], []]
epoch_loss_values = []
metric_values = []
metric_values_tc = []
metric_values_wt = []
metric_values_et = []
total_start = time.time()
for epoch in range(max_epochs):
epoch_start = time.time()
print("-" * 10)
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step_start = time.time()
step += 1
inputs, labels = (
batch_data["image"].to(device),
batch_data["label"].to(device),
)
optimizer.zero_grad()
with torch.cuda.amp.autocast():
outputs = model(inputs)
loss = loss_function(outputs, labels)
scaler.scale(loss).backward()
scaler.step(optimizer)
scaler.update()
epoch_loss += loss.item()
print(
f"{step}/{len(train_ds) // train_loader.batch_size}"
f", train_loss: {loss.item():.4f}"
f", step time: {(time.time() - step_start):.4f}"
)
lr_scheduler.step()
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
if (epoch + 1) % val_interval == 0:
model.eval()
with torch.no_grad():
for val_data in val_loader:
val_inputs, val_labels = (
val_data["image"].to(device),
val_data["label"].to(device),
)
val_outputs = inference(val_inputs)
val_outputs = [post_trans(i) for i in decollate_batch(val_outputs)]
dice_metric(y_pred=val_outputs, y=val_labels)
dice_metric_batch(y_pred=val_outputs, y=val_labels)
metric = dice_metric.aggregate().item()
metric_values.append(metric)
metric_batch = dice_metric_batch.aggregate()
metric_tc = metric_batch[0].item()
metric_values_tc.append(metric_tc)
metric_wt = metric_batch[1].item()
metric_values_wt.append(metric_wt)
metric_et = metric_batch[2].item()
metric_values_et.append(metric_et)
dice_metric.reset()
dice_metric_batch.reset()
if metric > best_metric:
best_metric = metric
best_metric_epoch = epoch + 1
best_metrics_epochs_and_time[0].append(best_metric)
best_metrics_epochs_and_time[1].append(best_metric_epoch)
best_metrics_epochs_and_time[2].append(time.time() - total_start)
torch.save(
model.state_dict(),
os.path.join(root_dir, "best_metric_model.pth"),
)
print("saved new best metric model")
print(
f"current epoch: {epoch + 1} current mean dice: {metric:.4f}"
f" tc: {metric_tc:.4f} wt: {metric_wt:.4f} et: {metric_et:.4f}"
f"\nbest mean dice: {best_metric:.4f}"
f" at epoch: {best_metric_epoch}"
)
print(f"time consuming of epoch {epoch + 1} is: {(time.time() - epoch_start):.4f}")
total_time = time.time() - total_start
print(f"train completed, best_metric: {best_metric:.4f} at epoch: {best_metric_epoch}, total time: {total_time}.")
train completed, best_metric: 0.7914 at epoch: 279, total time: 90155.70936012268.
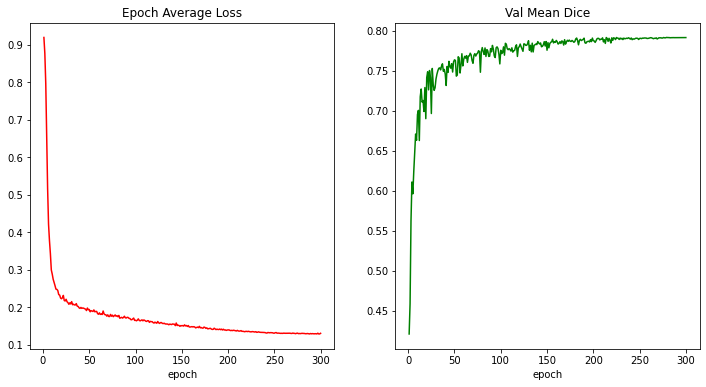
損失とメトリックのプロット
plt.figure("train", (12, 6))
plt.subplot(1, 2, 1)
plt.title("Epoch Average Loss")
x = [i + 1 for i in range(len(epoch_loss_values))]
y = epoch_loss_values
plt.xlabel("epoch")
plt.plot(x, y, color="red")
plt.subplot(1, 2, 2)
plt.title("Val Mean Dice")
x = [val_interval * (i + 1) for i in range(len(metric_values))]
y = metric_values
plt.xlabel("epoch")
plt.plot(x, y, color="green")
plt.show()
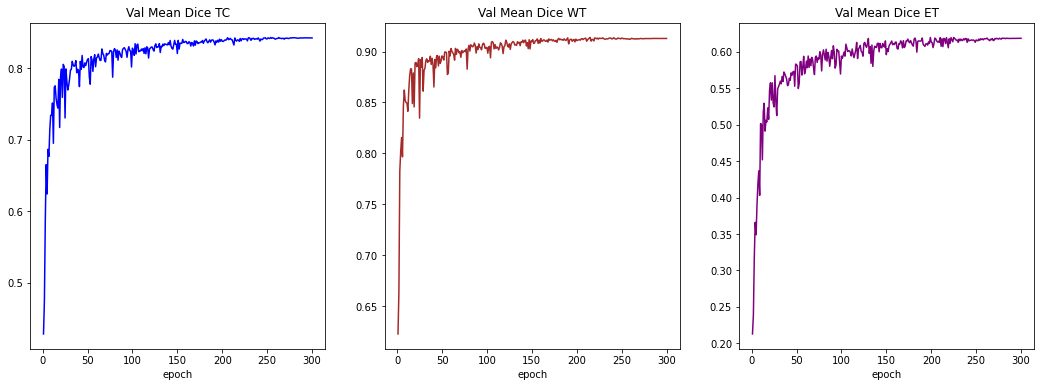
plt.figure("train", (18, 6))
plt.subplot(1, 3, 1)
plt.title("Val Mean Dice TC")
x = [val_interval * (i + 1) for i in range(len(metric_values_tc))]
y = metric_values_tc
plt.xlabel("epoch")
plt.plot(x, y, color="blue")
plt.subplot(1, 3, 2)
plt.title("Val Mean Dice WT")
x = [val_interval * (i + 1) for i in range(len(metric_values_wt))]
y = metric_values_wt
plt.xlabel("epoch")
plt.plot(x, y, color="brown")
plt.subplot(1, 3, 3)
plt.title("Val Mean Dice ET")
x = [val_interval * (i + 1) for i in range(len(metric_values_et))]
y = metric_values_et
plt.xlabel("epoch")
plt.plot(x, y, color="purple")
plt.show()
入力画像とラベルでベストモデル出力を確認する
model.load_state_dict(
torch.load(os.path.join(root_dir, "best_metric_model.pth"))
)
model.eval()
with torch.no_grad():
# select one image to evaluate and visualize the model output
val_input = val_ds[6]["image"].unsqueeze(0).to(device)
roi_size = (128, 128, 64)
sw_batch_size = 4
val_output = inference(val_input)
val_output = post_trans(val_output[0])
plt.figure("image", (24, 6))
for i in range(4):
plt.subplot(1, 4, i + 1)
plt.title(f"image channel {i}")
plt.imshow(val_ds[6]["image"][i, :, :, 70].detach().cpu(), cmap="gray")
plt.show()
# visualize the 3 channels label corresponding to this image
plt.figure("label", (18, 6))
for i in range(3):
plt.subplot(1, 3, i + 1)
plt.title(f"label channel {i}")
plt.imshow(val_ds[6]["label"][i, :, :, 70].detach().cpu())
plt.show()
# visualize the 3 channels model output corresponding to this image
plt.figure("output", (18, 6))
for i in range(3):
plt.subplot(1, 3, i + 1)
plt.title(f"output channel {i}")
plt.imshow(val_output[i, :, :, 70].detach().cpu())
plt.show()


元の画像 spacings 上の評価
val_org_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
EnsureChannelFirstd(keys=["image"]),
ConvertToMultiChannelBasedOnBratsClassesd(keys="label"),
Spacingd(keys=["image"], pixdim=(1.0, 1.0, 1.0), mode="bilinear"),
Orientationd(keys=["image"], axcodes="RAS"),
NormalizeIntensityd(keys="image", nonzero=True, channel_wise=True),
EnsureTyped(keys=["image", "label"]),
]
)
val_org_ds = DecathlonDataset(
root_dir=root_dir,
task="Task01_BrainTumour",
transform=val_org_transforms,
section="validation",
download=False,
num_workers=4,
cache_num=0,
)
val_org_loader = DataLoader(val_org_ds, batch_size=1, shuffle=False, num_workers=4)
post_transforms = Compose([
EnsureTyped(keys="pred"),
Invertd(
keys="pred",
transform=val_org_transforms,
orig_keys="image",
meta_keys="pred_meta_dict",
orig_meta_keys="image_meta_dict",
meta_key_postfix="meta_dict",
nearest_interp=False,
to_tensor=True,
),
Activationsd(keys="pred", sigmoid=True),
AsDiscreted(keys="pred", threshold_values=True),
])
model.load_state_dict(torch.load(
os.path.join(root_dir, "best_metric_model.pth")))
model.eval()
with torch.no_grad():
for val_data in val_org_loader:
val_inputs = val_data["image"].to(device)
val_data["pred"] = inference(val_inputs)
val_data = [post_transforms(i) for i in decollate_batch(val_data)]
val_outputs, val_labels = from_engine(["pred", "label"])(val_data)
dice_metric(y_pred=val_outputs, y=val_labels)
dice_metric_batch(y_pred=val_outputs, y=val_labels)
metric_org = dice_metric.aggregate().item()
metric_batch_org = dice_metric_batch.aggregate()
dice_metric.reset()
dice_metric_batch.reset()
metric_tc, metric_wt, metric_et = metric_batch[0].item(), metric_batch[1].item(), metric_batch[2].item()
print("Metric on original image spacing: ", metric)
print(f"metric_tc: {metric_tc:.4f}")
print(f"metric_wt: {metric_wt:.4f}")
print(f"metric_et: {metric_et:.4f}")
Metric on original image spacing: 0.7912478446960449 metric_tc: 0.8422 metric_wt: 0.9129 metric_et: 0.6187
データディレクトリのクリーンアップ
一時ディレクトリが使用された場合ディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
