MONAI 0.7 : tutorials : モジュール – 脾臓セグメンテーション・タスクの後処理変換 (翻訳/解説)
翻訳 : (株)クラスキャット セールスインフォメーション
作成日時 : 10/17/2021 (0.7.0)
* 本ページは、MONAI の以下のドキュメントを翻訳した上で適宜、補足説明したものです:
* サンプルコードの動作確認はしておりますが、必要な場合には適宜、追加改変しています。
* ご自由にリンクを張って頂いてかまいませんが、sales-info@classcat.com までご一報いただけると嬉しいです。

- 人工知能研究開発支援
- 人工知能研修サービス(経営者層向けオンサイト研修)
- テクニカルコンサルティングサービス
- 実証実験(プロトタイプ構築)
- アプリケーションへの実装
- 人工知能研修サービス
- PoC(概念実証)を失敗させないための支援
- テレワーク & オンライン授業を支援
- お住まいの地域に関係なく Web ブラウザからご参加頂けます。事前登録 が必要ですのでご注意ください。
- ウェビナー運用には弊社製品「ClassCat® Webinar」を利用しています。
◆ お問合せ : 本件に関するお問い合わせ先は下記までお願いいたします。
| 株式会社クラスキャット セールス・マーケティング本部 セールス・インフォメーション |
| E-Mail:sales-info@classcat.com ; WebSite: https://www.classcat.com/ ; Facebook |
MONAI 0.7 : tutorials : モジュール – 脾臓セグメンテーション・タスクの後処理変換
このノートブックは脾臓セグメンテーション・タスクのモデル出力に基づいて幾つかの後処理変換の使用方法を示します。
MONAI はモデル出力を処理するための後処理変換を提供しています。現在、変換は以下を含みます :
- Activations: 活性化層の追加 (Sigmoid, Softmax, etc.)。
- AsDiscrete: 離散値 (Argmax, One-Hot, Threshold 値等) に変換します。
- SplitChannel: マルチチャネル・データを複数のシングル・チャネルに分割する。
- KeepLargestConnectedComponent: セグメンテーション結果の輪郭を抽出します、これは元の画像へのマップに使用できてモデルを評価できます。
- LabelToContour: 接続コンポーネント分析に基づいてセグメンテーション・ノイズを除去する。
MONAI は同じデータ上で様々な前処理変換や後処理変換を適用して結果を結合するためのマルチ変換チェインをサポートしています、それはデータ辞書の指定された項目のコピーを作成する CopyItems 変換と想定される次元の指定項目を組み合わせるために ConcatItems 変換を提供し、そしてまたメモリを節約するために不要な項目を削除するための DeleteItems 変換も提供します。
典型的な使用方法は入力画像の 3 つの異なる強度範囲をスケールして結合することです :
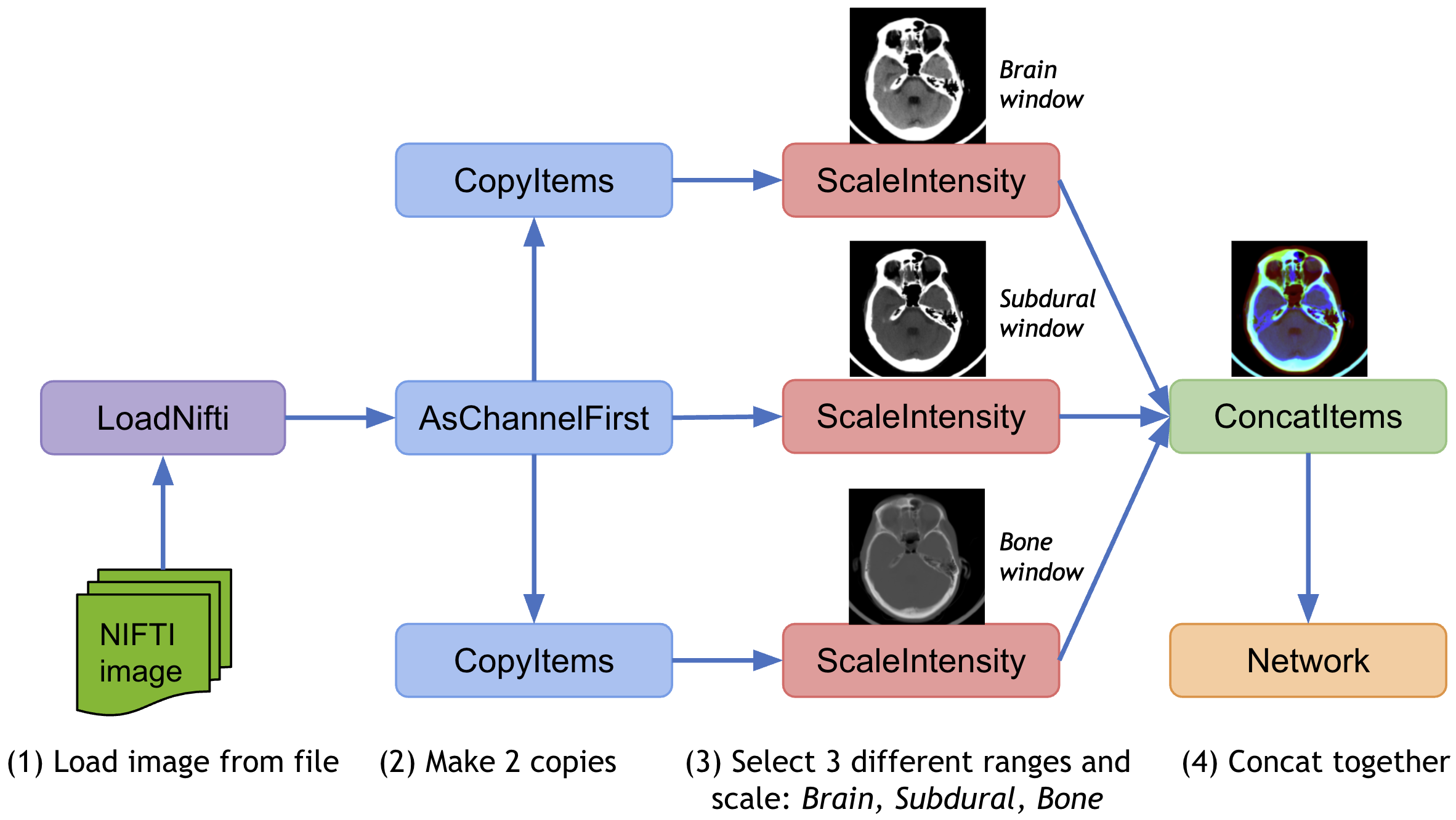
このチュートリアルは脾臓セグメンテーションのモデル出力に基づいて上の後処理変換の幾つかを示します。
環境のセットアップ
!python -c "import monai" || pip install -q "monai-weekly[gdown, nibabel, skimage, tqdm]"
!python -c "import matplotlib" || pip install -q matplotlib
%matplotlib inline
from monai.utils import set_determinism
from monai.transforms import (
AsDiscrete,
AddChanneld,
Compose,
CropForegroundd,
KeepLargestConnectedComponent,
LabelToContour,
LoadImaged,
Orientationd,
RandCropByPosNegLabeld,
ScaleIntensityRanged,
Spacingd,
EnsureTyped,
EnsureType,
)
from monai.networks.nets import UNet
from monai.networks.layers import Norm
from monai.metrics import DiceMetric
from monai.losses import DiceLoss
from monai.inferers import sliding_window_inference
from monai.data import CacheDataset, DataLoader, decollate_batch
from monai.config import print_config
from monai.apps import download_and_extract
import torch
import matplotlib.pyplot as plt
import tempfile
import shutil
import os
import glob
インポートのセットアップ
# Copyright 2020 MONAI Consortium
# Licensed under the Apache License, Version 2.0 (the "License");
# you may not use this file except in compliance with the License.
# You may obtain a copy of the License at
# http://www.apache.org/licenses/LICENSE-2.0
# Unless required by applicable law or agreed to in writing, software
# distributed under the License is distributed on an "AS IS" BASIS,
# WITHOUT WARRANTIES OR CONDITIONS OF ANY KIND, either express or implied.
# See the License for the specific language governing permissions and
# limitations under the License.
print_config()
MONAI version: 0.6.0rc1+23.gc6793fd0
Numpy version: 1.20.3
Pytorch version: 1.9.0a0+c3d40fd
MONAI flags: HAS_EXT = True, USE_COMPILED = False
MONAI rev id: c6793fd0f316a448778d0047664aaf8c1895fe1c
Optional dependencies:
Pytorch Ignite version: 0.4.5
Nibabel version: 3.2.1
scikit-image version: 0.15.0
Pillow version: 7.0.0
Tensorboard version: 2.5.0
gdown version: 3.13.0
TorchVision version: 0.10.0a0
ITK version: 5.1.2
tqdm version: 4.53.0
lmdb version: 1.2.1
psutil version: 5.8.0
pandas version: 1.1.4
einops version: 0.3.0
For details about installing the optional dependencies, please visit:
https://docs.monai.io/en/latest/installation.html#installing-the-recommended-dependencies
データディレクトリのセットアップ
MONAI_DATA_DIRECTORY 環境変数でディレクトリを指定できます。これは結果をセーブしてダウンロードを再利用することを可能にします。指定されない場合、一時ディレクトリが使用されます。
directory = os.environ.get("MONAI_DATA_DIRECTORY")
root_dir = tempfile.mkdtemp() if directory is None else directory
print(root_dir)
/workspace/data/medical
データセットのダウンロード
データセットをダウンロードして展開します。データセットは http://medicaldecathlon.com/ に由来します。
resource = "https://msd-for-monai.s3-us-west-2.amazonaws.com/Task09_Spleen.tar"
md5 = "410d4a301da4e5b2f6f86ec3ddba524e"
compressed_file = os.path.join(root_dir, "Task09_Spleen.tar")
data_dir = os.path.join(root_dir, "Task09_Spleen")
if not os.path.exists(data_dir):
download_and_extract(resource, compressed_file, root_dir, md5)
MSD 脾臓データセット・パスの設定
train_images = sorted(
glob.glob(os.path.join(data_dir, "imagesTr", "*.nii.gz")))
train_labels = sorted(
glob.glob(os.path.join(data_dir, "labelsTr", "*.nii.gz")))
data_dicts = [
{"image": image_name, "label": label_name}
for image_name, label_name in zip(train_images, train_labels)
]
train_files, val_files = data_dicts[:-9], data_dicts[-9:]
再現性のための決定論的訓練の設定
set_determinism(seed=0)
訓練と検証のための変換のセットアップ
train_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
AddChanneld(keys=["image", "label"]),
Spacingd(keys=["image", "label"], pixdim=(
1.5, 1.5, 2.0), mode=("bilinear", "nearest")),
Orientationd(keys=["image", "label"], axcodes="RAS"),
ScaleIntensityRanged(
keys=["image"], a_min=-57, a_max=164,
b_min=0.0, b_max=1.0, clip=True,
),
CropForegroundd(keys=["image", "label"], source_key="image"),
# randomly crop out patch samples from big image
# based on pos / neg ratio. the image centers
# of negative samples must be in valid image area
RandCropByPosNegLabeld(
keys=["image", "label"],
label_key="label",
spatial_size=(96, 96, 96),
pos=1,
neg=1,
num_samples=4,
image_key="image",
image_threshold=0,
),
EnsureTyped(keys=["image", "label"]),
]
)
val_transforms = Compose(
[
LoadImaged(keys=["image", "label"]),
AddChanneld(keys=["image", "label"]),
Spacingd(keys=["image", "label"], pixdim=(
1.5, 1.5, 2.0), mode=("bilinear", "nearest")),
Orientationd(keys=["image", "label"], axcodes="RAS"),
ScaleIntensityRanged(
keys=["image"], a_min=-57, a_max=164,
b_min=0.0, b_max=1.0, clip=True,
),
CropForegroundd(keys=["image", "label"], source_key="image"),
EnsureTyped(keys=["image", "label"]),
]
)
訓練と検証のための CacheDataset と DataLoader を定義する
train_ds = CacheDataset(
data=train_files, transform=train_transforms,
cache_rate=1.0, num_workers=4)
# train_ds = monai.data.Dataset(data=train_files, transform=train_transforms)
# use batch_size=2 to load images and use RandCropByPosNegLabeld
# to generate 2 x 4 images for network training
train_loader = DataLoader(train_ds, batch_size=2, shuffle=True, num_workers=4)
val_ds = CacheDataset(
data=val_files, transform=val_transforms, cache_rate=1.0, num_workers=4)
# val_ds = monai.data.Dataset(data=val_files, transform=val_transforms)
val_loader = DataLoader(val_ds, batch_size=1, num_workers=4)
100%|██████████| 32/32 [00:48<00:00, 1.51s/it] 100%|██████████| 9/9 [00:11<00:00, 1.32s/it]
モデル、損失、Optimizer を作成する
# standard PyTorch program style: create UNet, DiceLoss and Adam optimizer
device = torch.device("cuda:0")
model = UNet(
spatial_dims=3,
in_channels=1,
out_channels=2,
channels=(16, 32, 64, 128, 256),
strides=(2, 2, 2, 2),
num_res_units=2,
norm=Norm.BATCH,
).to(device)
loss_function = DiceLoss(to_onehot_y=True, softmax=True)
optimizer = torch.optim.Adam(model.parameters(), 1e-4)
dice_metric = DiceMetric(include_background=False, reduction="mean")
典型的な PyTorch 訓練プロセスを実行する
max_epochs = 160
val_interval = 2
best_metric = -1
best_metric_epoch = -1
epoch_loss_values = []
metric_values = []
post_pred = Compose([EnsureType(), AsDiscrete(argmax=True, to_onehot=True, num_classes=2)])
post_label = Compose([EnsureType(), AsDiscrete(to_onehot=True, num_classes=2)])
for epoch in range(max_epochs):
print("-" * 10)
print(f"epoch {epoch + 1}/{max_epochs}")
model.train()
epoch_loss = 0
step = 0
for batch_data in train_loader:
step += 1
inputs, labels = (
batch_data["image"].to(device),
batch_data["label"].to(device),
)
optimizer.zero_grad()
outputs = model(inputs)
loss = loss_function(outputs, labels)
loss.backward()
optimizer.step()
epoch_loss += loss.item()
print(
f"{step}/{len(train_ds) // train_loader.batch_size},"
f"train_loss: {loss.item():.4f}")
epoch_loss /= step
epoch_loss_values.append(epoch_loss)
print(f"epoch {epoch + 1} average loss: {epoch_loss:.4f}")
# validation progress
if (epoch + 1) % val_interval == 0:
model.eval()
with torch.no_grad():
for val_data in val_loader:
val_inputs, val_labels = (
val_data["image"].to(device),
val_data["label"].to(device),
)
roi_size = (160, 160, 160)
sw_batch_size = 4
val_outputs = sliding_window_inference(
val_inputs, roi_size, sw_batch_size, model)
val_outputs = [post_pred(i) for i in decollate_batch(val_outputs)]
val_labels = [post_label(i) for i in decollate_batch(val_labels)]
dice_metric(y_pred=val_outputs, y=val_labels)
metric = dice_metric.aggregate().item()
dice_metric.reset()
metric_values.append(metric)
if metric > best_metric:
best_metric = metric
best_metric_epoch = epoch + 1
torch.save(model.state_dict(),
"best_metric_model_post_transforms.pth")
print("saved new best metric model")
print(
f"current epoch: {epoch + 1} current mean dice: {metric:.4f}"
f"\nbest mean dice: {best_metric:.4f} "
f"at epoch: {best_metric_epoch}"
)
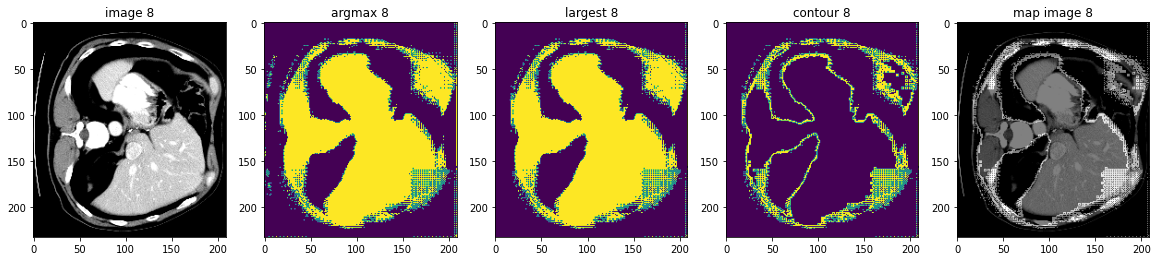
検証データセット上で後処理変換を実行する
ここでは AsDiscrete, KeepLargestConnectedComponent と LabelToContour をテストします。
model.load_state_dict(torch.load("best_metric_model_post_transforms.pth"))
model.eval()
with torch.no_grad():
for i, val_data in enumerate(val_loader):
roi_size = (160, 160, 160)
sw_batch_size = 4
val_data = val_data["image"].to(device)
val_output = sliding_window_inference(
val_data, roi_size, sw_batch_size, model)
# plot the slice [:, :, 80]
plt.figure("check", (20, 4))
plt.subplot(1, 5, 1)
plt.title(f"image {i}")
plt.imshow(val_data.detach().cpu()[0, 0, :, :, 80], cmap="gray")
plt.subplot(1, 5, 2)
plt.title(f"argmax {i}")
argmax = [AsDiscrete(argmax=True)(i) for i in decollate_batch(val_output)]
plt.imshow(argmax[0].detach().cpu()[0, :, :, 80])
plt.subplot(1, 5, 3)
plt.title(f"largest {i}")
largest = [KeepLargestConnectedComponent(applied_labels=[1])(i) for i in argmax]
plt.imshow(largest[0].detach().cpu()[0, :, :, 80])
plt.subplot(1, 5, 4)
plt.title(f"contour {i}")
contour = [LabelToContour()(i) for i in largest]
plt.imshow(contour[0].detach().cpu()[0, :, :, 80])
plt.subplot(1, 5, 5)
plt.title(f"map image {i}")
map_image = contour[0] + val_data[0]
plt.imshow(map_image.detach().cpu()[0, :, :, 80], cmap="gray")
plt.show()
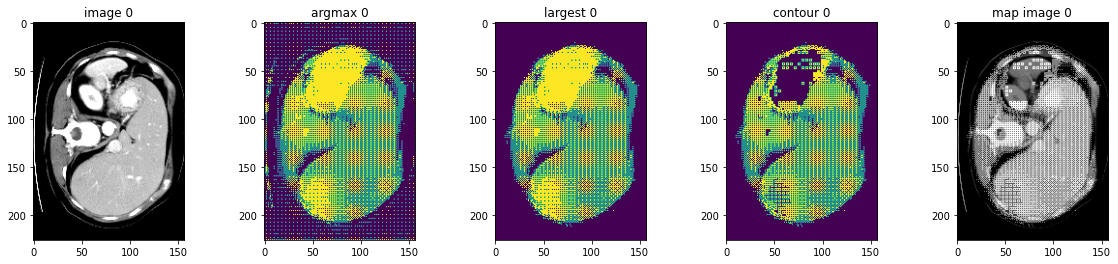
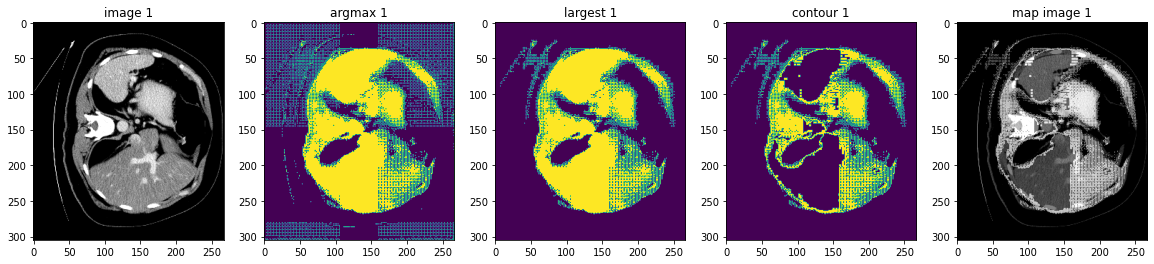
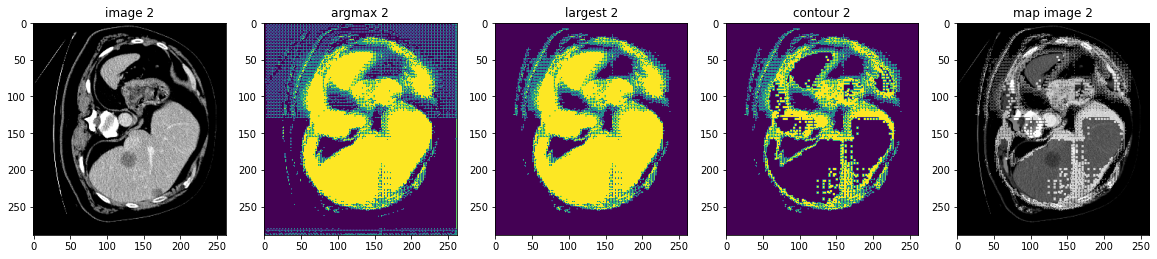
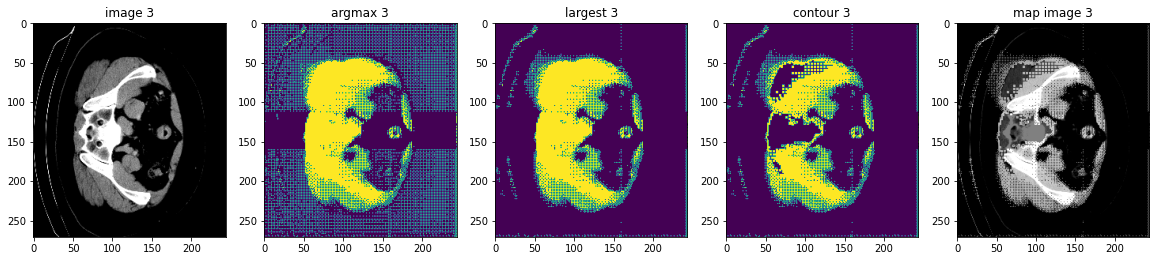
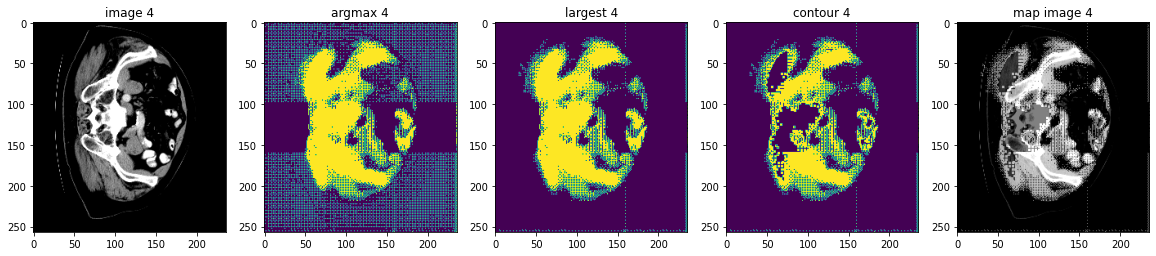

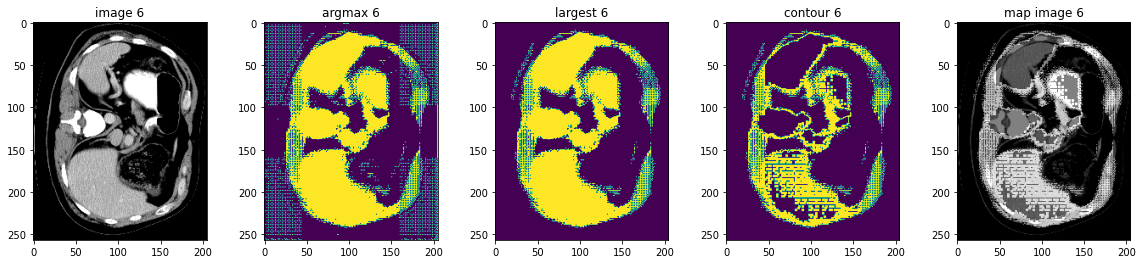
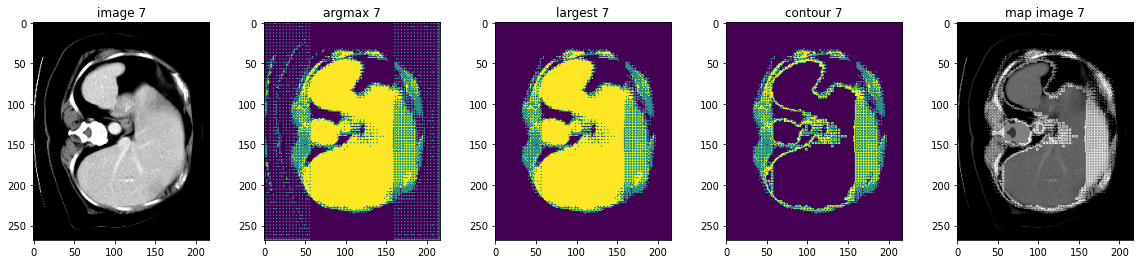
データディレクトリのクリーンアップ
一時ディレクトリが使用された場合にはディレクトリを削除します。
if directory is None:
shutil.rmtree(root_dir)
以上
